Mapeo cuantitativo de la deformación de un aneurisma de la aorta abdominal
Visión general
Fuente: Hannah L. Cebull1, Arvin H. Soepriatna1, John J. Boyle2 y Craig J. Goergen1
1 Weldon School of Biomedical Engineering, Universidad Purdue, West Lafayette, Indiana
2 Ingeniería Mecánica y Ciencia de los Materiales, Universidad de Washington en St. Louis, St Louis, Missouri
El comportamiento mecánico de los tejidos blandos, como los vasos sanguíneos, la piel, los tendones y otros órganos, está fuertemente influenciado por su composición de elastina y colágeno, que proporcionan elasticidad y fuerza. La orientación de la fibra de estas proteínas depende del tipo de tejido blando y puede variar desde una sola dirección preferida hasta intrincadas redes malladas, que pueden alterarse en el tejido enfermo. Por lo tanto, los tejidos blandos a menudo se comportan anisotrópicamente a nivel celular y de órganos, creando una necesidad de caracterización tridimensional. Desarrollar un método para estimar de forma fiable los campos de deformación unitaria dentro de tejidos o estructuras biológicas complejas es importante para caracterizar y comprender mecánicamente la enfermedad. La tensión representa cómo el tejido blando se deforma relativamente con el tiempo, y se puede describir matemáticamente a través de varias estimaciones.
La adquisición de datos de imagen a lo largo del tiempo permite estimar la deformación y la tensión. Sin embargo, todas las modalidades de imágenes médicas contienen cierta cantidad de ruido, lo que aumenta la dificultad de estimar con precisión la tensión in vivo. La técnica descrita aquí supera con éxito estos problemas mediante el uso de un método de estimación de deformación directa (DDE) para calcular campos de deformación unitaria 3D que varían espacialmente a partir de datos de imagen volumétrica.
Los métodos actuales de estimación de deformación unitaria incluyen correlación de imágenes digitales (DIC) y correlación de volumen digital. Desafortunadamente, DIC sólo puede estimar con precisión la tensión de un plano 2D, limitando severamente la aplicación de este método. Si bien son útiles, los métodos 2D como DIC tienen dificultadpara cuantificar la tensión en regiones que se someten a deformación 3D. Esto se debe a que el movimiento fuera del plano crea errores de deformación. La correlación de volumen digital es un método más aplicable que divide los datos de volumen inicialen regiones y encuentra la región más similar del volumen deformado, lo que reduce el error fuera del plano. Sin embargo, este método demuestra ser sensible al ruido y requiere suposiciones sobre las propiedades mecánicas del material.
La técnica que se muestra aquí elimina estos problemas mediante el uso de un método DDE, por lo que es muy útil en el análisis de datos de imágenes médicas. Además, es robusta a cepa alta o localizada. Aquí describimos la adquisición de datos de ultrasonido 4D cerrados y volumétricos, su conversión en un formato analizable y el uso de un código Matlab personalizado para estimar la deformación 3D y las cepas de Green-Lagrange correspondientes, un parámetro que describe mejor las grandes deformaciones. El tensor de cepa Green-Lagrange se implementa en muchos métodos de estimación de deformación unitaria 3D porque permite calcular F a partir de un ajuste de mínimos cuadrados (LSF) de los desplazamientos. La ecuación siguiente representa el tensor de cepa Green-Lagrange, E, donde F y I representan el gradiente de deformación y el tensor de identidad de segundo orden, respectivamente.
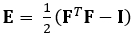 (1)
(1)
Principios
El ultrasonido 4D es un volumen dinámico que se adquiere utilizando un motor de traducción lineal unido a un transductor de ultrasonido, lo que permite la adquisición de bucles de vídeo secuenciales cerrados cardíacos y respiratorios a través de una región de interés. Este método es útil para visualizar estructuras complejas como el corazón, donde la hipertrofia o infarto causa geometrías únicas, o aneurismas aórticos, donde la expansión y disección de los vasos asimétricos a menudo ocurren en vasos tortuosos. Además, los datos 4D pueden proporcionar información espacial y temporal de alta resolución, que también es importante para las imágenes cardiovasculares.
El método DDE aplicado a los datos de ultrasonido 4D es superior a otros métodos porque utiliza el registro de imágenes no rígidas. Los tensores de gradiente de deformación se estiman tradicionalmente a partir de campos de desplazamiento después de la correlación de volumen digital. Por el contrario, el método DDE estima intrínsecamente los tensores de gradiente de deformación durante el registro del volumen optimizando una función de deformación que se elige cuidadosamente para ser directamente análoga al tensor de deformación. La función de deformación depende tanto de la posición espacial como del parámetro de deformación (p):
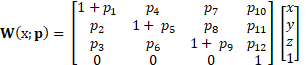 (2)
(2)
Los tres primeros elementos de esta función representan el tensor de gradiente de deformación, F, lo que permite que el cálculo de la deformación se incorpore directamente a la función de deformación. Este método de deformación se ha demostrado para aumentar la precisión y precisión de la estimación de la tensión en comparación con técnicas anteriores similares, ya que permite deformaciones grandes o localizadas que se encuentran comúnmente en los tejidos blandos.
Procedimiento
1. Configuración por ultrasonido 4D
- Cuando utilice el software de imágenes, utilice un portátil capaz de ejecutar software informático matemático para automatizar el proceso de adquisición 4D. Conecte el portátil con este código personalizado al sistema de ultrasonido a través del puerto USB. Tenga en cuenta que el software de imágenes tiene una función de ultrasonido 4D integrada en el software.
- Después de encender el sistema de ultrasonido, configure la unidad de monitoreo fisiológico mientras se asegura de que los botones de frecuencia cardíaca y temperatura estén encendidos. Inicializar la etapa del motor 3D conectada al soporte del transductor.
- Utilice el estadio y el transductor de ultrasonido adecuados para la adquisición de imágenes. Asegúrese de que se realizan todas las conexiones adecuadas.
- Proceda con la anestesia y preparación del animal para la toma de imágenes. Añadir pomada oftálmica a los ojos para evitar la desecación corneal, asegurar las patas a los electrodos del escenario, e insertar una sonda de temperatura rectal lubricada. Retire el pelaje en el área de interés usando una crema depilatoria.
- Asegúrese de que la crema depilatoria se haya completado. A continuación, aplicar una cantidad generosa de gel de transducción ultrasónica calentado al animal. Esto es especialmente importante para crear una buena conexión en toda la región de interés para las imágenes 4D.
2. Adquisición de ultrasonido sin ultrasonido 4D
- Comience un nuevo estudio sobre el sistema de ultrasonido y abra la ventana de imágenes en modo B (modo de brillo). Baje el transductor sobre el animal y localice la región de interés utilizando las perillas de los ejes X e Y en el escenario, asegurándose de que la frecuencia respiratoria no disminuya sustancialmente. Monitoree esto en la parte inferior de la pantalla.
- Coloque el transductor dentro del centro de la región de interés. A partir de ahí, se aproxima la distancia necesaria para que el transductor se mueva hacia arriba y hacia abajo de tal forma que se incluya toda la región de interés.
- Introduzca las dimensiones aproximadas en el código de software informático, incluido un tamaño de paso que suele ser de 0,08 mm para las imágenes de aneurisma de la aorta abdominal. Comience a ejecutar el código después de asegurarse de que el corazón del animal y las tasas respiratorias estén estables. Esto es importante para reducir los errores al reconstruir imágenes.
- Después de completar la adquisición de imágenes, exporte los datos como archivos XML sin procesar.
3. Conversión de datos de ultrasonido 4D
- Introduzca los archivos XML sin procesar en software que pueda convertir los datos en el formato adecuado para el análisis de manchas 3D. Aquí usamos Matlab para convertir archivos XML en archivos MAT. El script completo de Matlab está disponible aquí.
- Para una conversión adecuada, también será necesario introducir el número de fotogramas, el tamaño del paso y la resolución de salida deseada.
- Después de volver a muestrear la matriz a través del plano, importe el nuevo archivo MAT en el código de análisis de deformación unitaria 3D.
4. Análisis de código de tensión 3D
- Comience el análisis ajustando correctamente el archivo MAT importado. Por ejemplo, es posible que sea necesario cambiar la escala del volumen de la imagen para reducir el tiempo de cálculo.
- Introduzca la región que se va a analizar y determine la plantilla de malla adecuada para segmentar los datos de imagen como cuadros simples o polígonos elegidos manualmente. Es posible que sea necesario modificar el tamaño del cuadro de las regiones y el espaciado entre los puntos centrales para cada conjunto de datos. Los números óptimos elegidos para el tamaño del cuadro estarán alrededor del orden de píxeles de la entidad que se está rastreando, que se puede aproximar mirando el número de píxeles en dos dimensiones en un sector. El espaciado de las cajas determinará la resolución de los campos de deformación unitaria. Más cajas aumentarán la resolución, pero también pueden aumentar sustancialmente el tiempo de cálculo.
- Comience a calcular los jacobinos y gradientes iterativamente dentro de cada una de estas regiones. Una vez completado el precálculo, aplique la función de deformación.
- Calcule el tensor de gradiente de deformación. Primero calcule la deformación unitaria y, a continuación, calcule los valores propios y los autovectores utilizando el método de estimación de deformación directa.
- Trazar estos resultados en los planos deseados utilizando una técnica como la asignación de color de un plano de corte para representar el campo de deformación unitaria sobre la región de interés.
Resultados
Mediante el procedimiento descrito anteriormente, se adquirió una ecografía 4D de un aneurisma suprarrenal (AAA) de una angiotensina II. Se adquirieron varios bucles de vídeo EKV de eje corto a lo largo de la aorta y se combinaron para crear datos 4D, como se muestra en la Figura 1. Estos datos se convirtieron en un archivo MAT mediante un código personalizado, que luego se analizó en un código de cálculo de deformación unitaria 3D mediante una función de deformación. Después de optimizar los parámetros del código para un conjunto de datos específico, se produjo una vista representativa de eje largo con los valores de deformación unitaria correspondientes, así como un trazado de visualización de sectores 3D con un mapa de color de deformación unitaria superpuesta (Figura 2). Esta técnica DDE y los datos de deformación unitaria resaltan las variaciones espaciales heterogéneas en la tensión, especialmente cuando hay un trombo presente. Estos resultados se pueden correlacionar con la estructura del recipiente para determinar la relación entre la deformación in vivo y la composición del aneurisma.

Figura 1: Los bucles de visualización de kilohercios cerrados por ECG (EKV) de la aorta se adquieren de ubicaciones iniciales y finales introducidas manualmente, siguiendo un tamaño de paso de 0,2 mm.

Figura 2: Datos de ultrasonido de alta frecuencia 4D de un aneurisma de la aorta abdominal disuado murino representado en la sístole (A) con los principales campos de tensión estimados y superpuestos (B) (Scalebar a 5 mm). Vistas de eje largo y corto que representan las regiones aneurisma y sana que corresponden a la tensión principal en un ciclo cardíaco (sistolia: t-0,4) (C, D). Estos datos muestran niveles de tensión relativamente altos en regiones sanas y valores de tensión reducidos dentro del aneurisma de disección.
Aplicación y resumen
La caracterización mecánica localizada in vivo es una parte importante de la comprensión del crecimiento y la remodelación de los tejidos biológicos. En comparación con los enfoques existentes, el procedimiento de cuantificación de deformación unitaria descrito aquí utiliza un método mejorado para calcular con precisión la tensión 3D mediante la deformación óptima de la imagen no deformada antes de la correlación cruzada. Este método no utiliza ninguna suposición material para determinar las deformaciones unitarias dentro de los volúmenes de tejido. Desafortunadamente, la estimación de la tensión es confiable sólo hasta un tamaño de núcleo de 15x15x15 vóxeles cuando se utilizan datos de ultrasonido, lo que sugiere que este enfoque DDE puede no detectar características sutiles dentro de un campo de tensión. A pesar de esta limitación, sigue siendo una herramienta importante para investigar las respuestas mecánicas, diagnosticar la patología y mejorar los modelos de enfermedades.
Muchas áreas de investigación más allá de los aneurismas aórticos pueden beneficiarse de esta herramienta de medición de tensión. La tensión cardíaca también se puede cuantificar fácilmente con este método. Debido a que el miocardio sufre deformación 3D durante el ciclo cardíaco, cuantificar la tensión en tres dimensiones es integral para caracterizar de forma fiable la dinámica de este tejido. Los datos fiables de las cepas son especialmente importantes cuando se realiza un seguimiento de la progresión de la enfermedad en modelos animales.
El análisis de deformación unitaria 3D también se puede aplicar a las imágenes por ultrasonido intestinal. La caracterización mecánica del tejido intestinal se lleva a cabo más comúnmente in vitro. Sin embargo, esto no siempre es una verdadera representación del comportamiento real de los intestinos in vivo debido a los efectos de las estructuras circundantes. Como ejemplo de traducción clínica de este enfoque, el cálculo de la tensión a partir de imágenes de fibrosis intestinal debido a una presión luminal anormal podría proporcionar detección temprana de áreas problemáticas que requieren intervención quirúrgica.
Más allá de las aplicaciones a mayor escala, este método también se puede aplicar al nivel celular mediante el uso de técnicas de imagen de mayor resolución, como la microscopía confocal. Caracterizar la matriz extracelular es importante para entender cómo se comunican las células. Se ha llevado a cabo mucha investigación sobre la caracterización bioquímica, pero entender cómo se puede llevar a cabo la comunicación a través de respuestas mecánicas requiere una comprensión de la deformación y la tensión. La tensión a granel no es beneficiosa porque no hay manera de determinar el origen del cambio de deformación. La aplicación de un enfoque DDE de alta resolución podría revelar directamente cómo la matriz extracelular responde a los cambios mecánicos.
Agradecimientos
Nos gustaría reconocer a John Boyle, Guy Genin y Stavros Thomopoulos por la contribución del código Matlab personalizado DDE capaz de estimar directamente la cepa Lagrange-Green.
Saltar a...
Vídeos de esta colección:

Now Playing
Mapeo cuantitativo de la deformación de un aneurisma de la aorta abdominal
Biomedical Engineering
4.6K Vistas

Obtención de imágenes de muestras biológicas con microscopía óptica y confocal
Biomedical Engineering
35.8K Vistas

Imágenes con MEB de muestras biológicas
Biomedical Engineering
23.8K Vistas

Biodistribución de nanotransportadores de fármacos: Aplicaciones del MEB
Biomedical Engineering
9.3K Vistas

Imágenes por ultrasonido de alta frecuencia de la aorta abdominal
Biomedical Engineering
14.6K Vistas

Tomografía fotoacústica para obtener imágenes de sangre y lípidos de la Aorta Infrarrenal
Biomedical Engineering
5.7K Vistas

Resonancia Magnética Cardíaca
Biomedical Engineering
14.8K Vistas

Simulaciones de dinámica de fluidos computacionales del flujo sanguíneo en un aneurisma cerebral
Biomedical Engineering
11.8K Vistas

Imágenes de fluorescencia infrarroja de aneurismas aórticos abdominales
Biomedical Engineering
8.3K Vistas

Técnicas no invasivas para medir la presión arterial
Biomedical Engineering
11.9K Vistas

Adquisición y análisis de una señal de ECG (electrocardiografía)
Biomedical Engineering
106.2K Vistas

Resistencia a la tracción de biomateriales reabsorbibles
Biomedical Engineering
7.5K Vistas

Imágenes por micro-CT de la médula espinal de un ratón
Biomedical Engineering
8.0K Vistas

Visualización de la degeneración articular de rodilla posterior a una lesión no invasiva del LCA en ratas
Biomedical Engineering
8.2K Vistas

Imágenes combinadas de SPECT y TC para visualizar la funcionalidad cardíaca
Biomedical Engineering
11.0K Vistas
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados