Method Article
Anwendung der Chemieinformatik zur Entwicklung einer strukturdurchsuchbaren Datenbank analytischer Methoden
In diesem Artikel
Zusammenfassung
Dieser Artikel beschreibt AMOS, die webbasierte Datenbank für analytische Methoden und offene Spektren, eine Chemieinformatik-Anwendung, die Forschern einen einfachen Zugang zu Analysemethoden und Spektraldaten ermöglicht.
Zusammenfassung
Die Analysemethoden können von detaillierten regulatorischen Dokumenten bis hin zu einfacheren Zusammenfassungen reichen. Regulatorische Methoden können Informationen über zugängliche Analyten, unterstützte Matrices, erforderliche Reagenzien, statistische Leistung, Ringversuchsvalidierung und andere Besonderheiten umfassen. Zusammenfassungen bieten in der Regel einen allgemeinen Überblick über Reagenzien, Instrumentierung und oft eine kurze Liste von Analyten. Analysemethoden von US-Regierungsbehörden, darunter die U.S. Environmental Protection Agency (USEPA), U.S. Geological Survey (USGS), U.S. Department of Agriculture (USDA), die Food and Drug Administration (FDA) und andere, bieten detaillierte Verfahrensinformationen. Gerätehersteller wie Agilent, Shimadzu, Thermo Fisher Scientific, Sciex und andere bieten ebenfalls Zugriff auf Hunderte von Anwendungshinweisen, die als zusammenfassende Methoden betrachtet werden können. Im Rahmen dieser Studie wurde eine cheminformatgestützte Datenbank von Methoden entwickelt, in denen Chemikalien aus Methodendokumenten extrahiert werden, wobei Identifikatoren (Namen und/oder Registrierungsnummern des Chemical Abstracts Service (CASRN)) auf chemische Strukturen abgebildet werden. Die daraus resultierende Datenbank mit ca. 7.000 Methoden ist nach Identifikator, chemischer Struktur und struktureller Ähnlichkeit durchsuchbar und wird durch ca. eine Million Public Domain-Spektren (LC/MS, GC/MS, NMR und IR) ergänzt. Die Anwendung unterstützt die Suche nach analytischen Methoden und die Filterung basierend auf Analyten, funktionaler Verwendung, Methodenquellen und anderen verwandten Metadaten.
Einleitung
Beispiele für die webbasierte Bereitstellung von Chemiedaten für die Community sind Anwendungen wie PubChem1, ChemSpider2 und das CompTox Chemicals Dashboard (CCD)3. Es wurden Anstrengungen unternommen, um Details zu Analysemethoden in Umlauf zu bringen, die in Zeitschriftenartikeln veröffentlicht wurden, von Geräteherstellern als technische Anwendungshinweise veröffentlicht wurden, von Regierungsbehörden als Standardarbeitsanweisungen oder Regulierungsmethoden zur Verfügung gestellt und von Normungsorganisationen wie der Internationalen Organisation für Normung (ISO) herausgegeben wurden. Zehntausende von Chemikalien wurden von diesen Quellen unter einer Vielzahl von Bedingungen und Analysetechniken untersucht. Dieser umfangreiche Quellenkatalog deckt eine Vielzahl von Substanzen ab und umfasst Szenarien, die von der Quantifizierung einer einzelnen Chemikalie in einer bestimmten Matrix (z. B. Blut) über Mischungen von Pestiziden und deren Rückständen in bestimmten Kulturen bis hin zu Hunderten von im Trinkwasser identifizierten Chemikalien reichen. Während viele Analysemethoden über öffentliche Suchmaschinen gefunden werden können, sind nicht alle frei verfügbar oder frei zugänglich.
Das Auffinden bestimmter Informationen von Interesse kann eine Herausforderung sein. Allzweck-Suchmaschinen sind nicht für Chemiedaten optimiert, und ihre Ranking-Algorithmen können qualitativ hochwertige Inhalte verschleiern, die für ein enges Publikum gedacht sind. Die Suche auf Zeitschriften-Websites kann zu gezielteren Ergebnissen führen, aber der Zugang ist oft eingeschränkt, da nur Abstracts öffentlich zugänglich sind, was es schwierig macht, den Nutzen einer Methode zu beurteilen. Darüber hinaus werden kritische Parameter wie Probenmatrizen, Nachweisgrenzen und Quantifizierung oft nicht in einem strukturierten Format gespeichert. Eine weitere große Herausforderung liegt in der Variation und Inkonsistenz von chemischen Identifikatoren, Namen und Synonymen, die mit einer einzigen Chemikalie verbunden sind. Der Mangel an strukturierten Methodendaten schränkt die Entwicklung von Software-Tools ein, die jahrzehntelang angesammeltes Wissen über analytische Chemie und damit verbundene Veröffentlichungen nutzen könnten.
Infolge dieser Herausforderungen und Einschränkungen besteht ein Bedarf an einer kuratierten, chemieorientierten Anwendung zur Harmonisierung und Suche nach analytischen Methoden, die anderswo nicht identifiziert wurde. Um diese Lücke zu schließen, hat die U.S. Environmental Protection Agency AMOS entwickelt, die Datenbank Analytical Methods and Open Spectra sowie eine webbasierte Anwendung. AMOS sammelt und organisiert derzeit drei Arten von Datensätzen: analytische Methoden, verschiedene analytische Spektren und eine breite Kategorie von ergänzenden Dokumenten, die zusammen als Fact Sheets bezeichnet werden. Jeder Datensatz ist mit den chemischen Zielanalyten und -reagenzien der Methode verknüpft. Die Daten können auf verschiedene Weise durchsucht werden, z. B. nach Textabfragen, chemischer Struktur und struktureller oder spektraler Ähnlichkeit.
Die AMOS-Anwendung konzentriert sich in erster Linie auf die Bereitstellung von Open Access und Open Data-Datensätzen. Wenn möglich, werden Datensätze in der Datenbank mit Hyperlinks zu ihren ursprünglichen Quellen versehen. Datensätze, die nicht unter einer offenen Lizenzierung stehen und daher nicht direkt in der Datenbank gespeichert sind, können trotzdem eingebunden und per URL abgerufen werden, sofern sie anderweitig verfügbar sind. Dies gilt für zwei Arten von Datensätzen: analytische Methoden, die sich hinter Bezahlschranken befinden, typischerweise von Zeitschriften oder Standardorganisationen, zu denen die EPA Zugang hat, und Spektren, die verfügbar sind, aber einen Login-Zugang erfordern.
Datenquellen unterscheiden sich in der Art und Weise, wie Datensätze strukturiert sind, was einen erheblichen Aufwand bei der Extraktion und Kuration erfordert, um den Inhalt zusammenzustellen und zu harmonisieren. Die meisten Datensätze enthalten Stoffidentifikatoren (z. B. CASRN, DTXSID, InChIKey, gebräuchliche Namen), und in vielen Fällen ist die Extraktion unkompliziert. Die Zuordnung dieser Identifikatoren zu chemischen Strukturen und Stoffdetails kann jedoch komplex sein. Einige Identifikatoren können direkt mit Einträgen in der DSSTox-Datenbank (Distributed Structure-Searchable Toxicity) der EPA abgeglichen werden4; Wenn keine Übereinstimmungen gefunden werden, werden Identifikatoren mit bestehenden Stoffen verknüpft oder neue Stoffe registriert. Die AMOS-Initiative hat daher zur Erweiterung der DSSTox-Datenbank geführt und die grundlegenden Daten zur Unterstützung anderer EPA-Datenbanken und -Anwendungen, wie z. B. des CompTox Chemicals Dashboard3, verbessert.
Für bestimmte wertvolle Zusatzinformationen ist eine manuelle Kuration erforderlich. Bei analytischen Methoden sind experimentelle Parameter wie Nachweis- und Quantifizierungsgrenzen, Probenmatrix und Analysemethodik nicht standardisiert organisiert, und automatisierte Werkzeuge können diese Informationen aufgrund ihrer inkonsistenten Speicherung nicht identifizieren.
Zwei Elemente der Aufzeichnungsinformationen, die mit der Probe verbundenen Medien und die funktionelle Verwendung des Analyten, sind für die laufenden Bemühungen zur Überwachung von Gefahren und Expositionsbedenken durch Kontaminanten von großer Bedeutung. Daher wurde der Strukturierung dieser Attribute innerhalb der Datensatzdaten große Aufmerksamkeit geschenkt. Für dieses Projekt wurde eine Ontologie von funktionalen Nutzungsklassifikationen entwickelt. Diese Ontologie organisiert die funktionalen Verwendungen von Substanzen in einer hierarchischen Struktur, die von allgemeineren "übergeordneten" Verwendungen bis hin zu spezifischeren "untergeordneten" Verwendungen reicht. Die Ontologie erleichtert die Erforschung von Stoffen aus der Anwendungsperspektive und unterstützt Forschungsinitiativen, die die funktionelle Verwendung als Mittel zur Bewertung von Exposition und Gefahr betonen 5,6. Darüber hinaus wurden die Methoden nach der harmonisierten Medienkategorie ihrer Stichproben gekennzeichnet, wie sie in der Multimedia-Monitoring-Datenbank (MMDB)7 der EPA spezifiziert ist. Diese Kategorisierung ermöglicht die Suche nach Chemikalien auf der Grundlage ihres Vorkommens in bestimmten Medien und rationalisiert die Entwicklung von Lösungen, die sich auf den Nachweis von Chemikalien in bestimmten Umwelt- oder biologischen Proben konzentrieren. Diese Annotationen verbessern die Integration von AMOS in expositions- und gefahrenorientierte Arbeitsabläufe, die innerhalb der EPA entwickelt werden.
Bei der Zusammenstellung der Spektren erfordert die Herausforderung der Verarbeitung verschiedener Dateiformate - von denen einige nur nominell standardisiert sind - und des Parsens der begleitenden Metadaten oft eine benutzerdefinierte Handhabung. In Fällen, in denen Spektralsammlungen mit einer Publikation verknüpft sind, müssen die in der Publikation dokumentierten Details möglicherweise manuell extrahiert werden, um die Daten zu laden. Diese Bemühungen haben zu einer Datenbank geführt, die diese unterschiedlichen Spektren integriert und strukturiert, so dass die Forscher bei zukünftigen Unternehmungen keine aufwändige Kuratierung mehr leisten müssen.
Mit Stand März 2025 enthält die Datenbank etwa 935.000 Spektren, wobei fast 99 % Massenspektren und kleinere Sammlungen von NMR (~2.000) und IR (~400) sind. Darüber hinaus gibt es ca. 770.000 extern verlinkte Spektren (verbunden mit der SpectraBase-Datenbank8), ~36.000 Factsheets und ~7.400 analytische Methoden. Die in die Anwendung integrierten Substanzen sind eine Teilmenge derjenigen aus der DSSTox-Datenbank, die in das CompTox Chemicals Dashboard (CCD) eingebunden ist und über 1,2 Millionen Substanzen enthält.
Protokoll
Der Großteil der AMOS-Funktionalität kann in drei Kategorien unterteilt werden: Suche nach Datensätzen für bestimmte Substanzen, Suche nach bestimmten Stoffsammlungen oder Suche zwischen Kategorien von Datensätzen. Die einzelnen Seiten für diese Funktionen können alle über die Navigationsleiste oben auf jeder Seite aufgerufen werden. Die Anwendung wird derzeit über das AMOS-Modul auf https://hcd.rtpnc.epa.gov/#/ bereitgestellt. Die in dieser Studie verwendeten Softwaretools sind in der Materialtabelle aufgeführt.
1. Suche nach Datensätzen für bestimmte Stoffe
- Allgemeine Suche: Führen Sie eine allgemeine Suche durch, um eine Liste aller Arten von Datensätzen zu erhalten, die mit einem einzelnen Stoff verbunden sind (siehe Abbildung 1).
- Geben Sie entweder in das Textfeld oben links in der Navigationsleiste oder in das Suchfeld auf der Startseite einen Stoffnamen, CASRN, InChIKey oder DSSTox-Stoffidentifikator (DTXSID) ein. Drücken Sie die Eingabetaste oder klicken Sie auf Suchen , um die Suche auszuführen.
HINWEIS: Die Suchleiste auf der Startseite verfügt über eine zusätzliche Option zur Suche nach Teilzeichenfolgen. Weitere Informationen finden Sie im Abschnitt über die Suche nach partiellen Kennungen (Schritt 2.2). - Wenn der gesuchte Identifikator erkannt wird und mit einem einzelnen Stoff übereinstimmt, werden auf der linken Seite der Seite einige grundlegende Informationen über den Stoff und eine Tabelle mit allen Datensätzen angezeigt, die mit diesem Stoff verknüpft sind. Wählen Sie eine Zeile in dieser Tabelle aus, um den zugeordneten Datensatz auf der rechten Seite der Seite anzuzeigen, wenn er direkt in der Datenbank gespeichert ist.
- Wenn der gesuchte Identifikator mit mehreren Stoffen übereinstimmt - z. B. mit einer Abkürzung, die für mehr als einen Stoff verwendet wird - wird eine Begriffsklärungsaufforderung angezeigt, in der der Benutzer auswählen kann, welchen Stoff er sehen möchte. Wählen Sie einen Stoff aus dieser Liste aus, und er wird zur Anzeige eines erkannten Stoffes weitergeleitet.
- Um die Ergebnistabelle zu filtern, klicken Sie auf die Registerkarten direkt über der Tabelle, um nach Datensatztyp zu filtern (dadurch werden auch verschiedene Spalten ein- und ausgeblendet), geben Sie Text in die Felder oben in der Tabelle ein, um nach anderen Aspekten der Daten zu filtern, und aktivieren Sie die Kontrollkästchen über den Registerkarten, um nach breiteren Eigenschaften der Daten zu filtern.
- Geben Sie entweder in das Textfeld oben links in der Navigationsleiste oder in das Suchfeld auf der Startseite einen Stoffnamen, CASRN, InChIKey oder DSSTox-Stoffidentifikator (DTXSID) ein. Drücken Sie die Eingabetaste oder klicken Sie auf Suchen , um die Suche auszuführen.
- Batch-Suche: Führen Sie eine Batch-Suche durch, um eine Tabellenkalkulationsdatei zu generieren und herunterzuladen, die Informationen zu allen Datensätzen in der Datenbank auflistet, die mit einer bestimmten Liste von Stoffen verknüpft sind. (siehe Abbildung 2).
- Geben Sie im Eingabedatenfeld eine Liste der zu durchsuchenden DTXSIDs ein, eine pro Zeile. Wenn DTXSIDs nicht verfügbar sind, verwenden Sie den Link auf der Seite, um zu einem CCD-Tool zu navigieren, das DTXSIDs mit anderen IDs bereitstellen kann.
- Verwenden Sie die Kontrollkästchen unter Suchoptionen , um Ergebnisse herauszufiltern oder zusätzliche Informationen an Datensätze anzuhängen. Die Optionen sind in fünf Kategorien unterteilt: Filtern nach Datensatztypen, Filtern nach Analysemethoden, Anhängen zusätzlicher Informationen auf Stoffebene an die Ergebnisdatei, Anhängen zusätzlicher Informationen auf Datensatzebene (derzeit nur für Massenspektren verfügbar) und einige verschiedene Optionen.
HINWEIS: Optionen mit einer gestrichelten Unterstreichung verfügen über einen Text, in dem die Option ausführlicher erläutert wird. Bewegen Sie den Mauszeiger über die Beschriftung der Option, um sie anzuzeigen. - Klicken Sie unten auf der Seite auf Suchen , um die Suche auszuführen.
HINWEIS: Die Ausgabetabelle enthält eine Liste der Stoff-Datensatz-Zuordnungen zusammen mit Stoffidentifikatoren, Quellenlinks und einigen anderen grundlegenden Informationen. Wenn mehrere gesuchte Stoffe in einem Datensatz erscheinen, wird der Datensatz für jeden Stoff einmal angezeigt.
- Strukturelle Ähnlichkeitssuche: Führen Sie diese Suche durch, um Listen von Methoden und Datenblättern in der Datenbank zu erhalten, die entweder den gesuchten Stoff oder einen Stoff mit einem ausreichend hohen Tanimoto-Strukturähnlichkeitskoeffizienten enthalten (siehe Abbildung 3).
HINWEIS: Diese Suche kann in Fällen nützlich sein, in denen ein Stoff von Interesse in keinem Verfahren vorkommt, aber Methoden mit sehr ähnlichen Stoffen möglicherweise als Referenz verwendet werden könnten.- Geben Sie einen DTXSID-, InChIKey-, CASRN- oder Stoffnamen in das Suchfeld ein und klicken Sie auf Suchen oder drücken Sie die Eingabetaste. Die Suche kann 20-30 Sekunden dauern.
- Sobald die Suche abgeschlossen ist, wird unten eine Tabelle mit Registerkarten angezeigt. Wählen Sie eine Registerkarte aus, um die Ergebnisse der Suche zu durchsuchen.
- Die ersten beiden Registerkarten listen die gefundenen Methoden und Informationsblätter auf. Wählen Sie eine Option aus, um eine Ansicht dieses Dokuments auf der rechten Seite der Seite anzuzeigen. Methoden oder Informationsblätter, die den gesuchten Stoff enthalten, sind fett gedruckt.
- Auf der dritten Registerkarte sind ähnliche Stoffe aufgeführt, die in Methoden oder Informationsblättern gefunden wurden. Wählen Sie eine Zeile in der Tabelle aus, um einen Vergleich zwischen dem gesuchten Stoff und dem in der Tabelle ausgewählten Stoff aufzurufen. Wenn die gesuchte Substanz selbst in irgendwelchen Dokumenten gefunden wurde, wird sie fett gedruckt.
- Verwenden Sie die Auswahl für die minimale Stoffähnlichkeit filtern oben, um Ergebnisse aus der Suche auszublenden, bei denen Substanzen unterhalb des ausgewählten Ähnlichkeitsschwellenwerts fehlen.

Abbildung 1: Suchergebnisse für Datensätze, die Cholesterin enthalten. Eine allgemeine Suche nach "Cholesterin" zeigt eine Liste mit übereinstimmenden Datensätzen in der Tabelle (links). Auf der rechten Seite wird das Massenspektrum einer ausgewählten Schallplatte angezeigt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 2: Schnittstelle für die Batch-Suche. Das Suchfeld enthält zwei Stoffe, die durch ihre DTXSIDs identifiziert werden. Für die Abfrage werden Standardsuchoptionen ausgewählt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 3: Struktur der Suchergebnisse für 1P-LSD. In der Tabelle sind Methoden aufgeführt, die strukturell ähnliche Stoffe enthalten. Auf der rechten Seite wird eine ausgewählte Methode angezeigt. Keine fett formatierten Einträge in der Tabelle weisen darauf hin, dass 1P-LSD in keiner der aufgeführten Methoden vorkommt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
2. Suche nach Substanzen
- ClassyFire-Suche: Führen Sie diese Suche durch, um alle Stoffe aufzulisten, die zu den angegebenen ersten vier Stufen einer ClassyFire-Klassifizierung9 gehören (siehe Abbildung 4).
- Wählen Sie mithilfe der vier Felder oben auf der Seite nacheinander die obersten vier Ebenen der Klassifizierung aus. Nachdem Sie jede der ersten drei ausgewählt haben, verwenden Sie die Schaltfläche unter diesem Feld, um die Liste der Klassifizierungen eine Ebene tiefer abzurufen. Für die vierte Schaltfläche führt die Schaltfläche unten die Suche aus.
HINWEIS: Sobald die Suche abgeschlossen ist, wird die folgende Tabelle durch eine Liste der Stoffe ergänzt, die unter dieser Einstufung vorhanden sind. Die Tabelle enthält gängige Identifikatoren und Stoffinformationen sowie die Anzahl der in AMOS vorhandenen Datensätze. - Verwenden Sie die Schaltflächen zwischen der Klassenauswahl und der Tabelle, um vier Funktionsbits zuzulassen:
- Klicken Sie auf Klassifizierung in URL kopieren , um eine URL in die Zwischenablage zu kopieren, die, wenn sie in einem neuen Browser-Tab oder -Fenster geladen wird, automatisch die Klassifizierungsebenen vorausfüllt und die Suche ausführt.
- Klicken Sie auf Auswahl zurücksetzen , um die Auswahl in den Klassifizierungsfeldern zurückzusetzen. Die Tabelle der gefundenen Substanzen wird nicht zurückgesetzt.
- Klicken Sie auf Tabelle herunterladen , um den Download einer Tabellenkalkulationsdatei aufzufordern, die alle sichtbaren Felder und Datensätze in der Tabelle enthält, mit Ausnahme der Stoffbilder. Wenn die Filter oben in der Ergebnistabelle verwendet werden, werden die heruntergeladenen Ergebnisse ebenfalls gefiltert, aber der Inhalt der Filter wird nicht eingeschlossen.
- Klicken Sie auf Ausgewählte Substanzen an Batch-Suche senden , um einen neuen Tab für die Batch-Suche zu öffnen, wobei das Feld für die Auflistung von DTXSIDs mit den aus den ClassyFire-Suchergebnissen ausgewählten Substanzen vorausgefüllt ist. Die Auswahl einzelner Substanzen kann mit dem Kontrollkästchen in jeder Zeile erfolgen; Die Auswahl oder Abwahl aller Stoffe kann durch Anklicken des Kontrollkästchens in der Kopfzeile der Tabelle erfolgen. Siehe Schritt 1.2 für Details zur Batch-Suche.
- Wählen Sie mithilfe der vier Felder oben auf der Seite nacheinander die obersten vier Ebenen der Klassifizierung aus. Nachdem Sie jede der ersten drei ausgewählt haben, verwenden Sie die Schaltfläche unter diesem Feld, um die Liste der Klassifizierungen eine Ebene tiefer abzurufen. Für die vierte Schaltfläche führt die Schaltfläche unten die Suche aus.
- Suche nach partiellen Identifikatoren: Führen Sie diese Option durch, um alle Stoffe zu finden, die mit einem nicht eindeutigen Identifikator übereinstimmen (siehe Abbildung 5). Die aktuellen Optionen sind die Namensteilzeichenfolge (die sowohl den von der EPA bevorzugten Namen als auch gebräuchliche Synonyme abdeckt), der erste InChIKey-Block, die genaue Summenformel und eine Reihe von monoisotopischen Massen.
- Wählen Sie oben auf der Seite eine Kennung aus und geben Sie die Informationen in die angrenzenden Felder ein.
- Klicken Sie auf Suchen , um die Suche auszuführen.
- Wenn die Suche abgeschlossen ist, wird die Tabelle mit einer Liste von Stoffen gefüllt, die mit dem partiellen Identifikator übereinstimmen, sowie mit Informationen darüber, wie oft sie in der AMOS-Datenbank und in anderer Literatur vorkommen. Verwenden Sie die Filter oben in den Spalten der Tabelle, um die Ergebnisse weiter zu verfeinern, und verwenden Sie das Kontrollkästchen Mehrkomponenten-Stoffe anzeigen , um Stoffe, die aus mehreren Verbindungen bestehen, ein- oder auszublenden.
HINWEIS: Wenn eine Suche nach Namensteilzeichenfolgen ausgeführt wurde, wird eine Spalte mit den gefundenen Synonymen angezeigt. Wenn ein Stoff nur über Synonyme gefunden wird, d.h. wenn der bevorzugte Name den Teilstring nicht enthält, wird der bevorzugte Name kursiv geschrieben.

Abbildung 4: Suchergebnisse für die ClassyFire-Klassifizierung. Zu den Ergebnissen gehören Informationen auf Stoffebene und die Anzahl der Datensätze pro Einstufungsgruppe. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
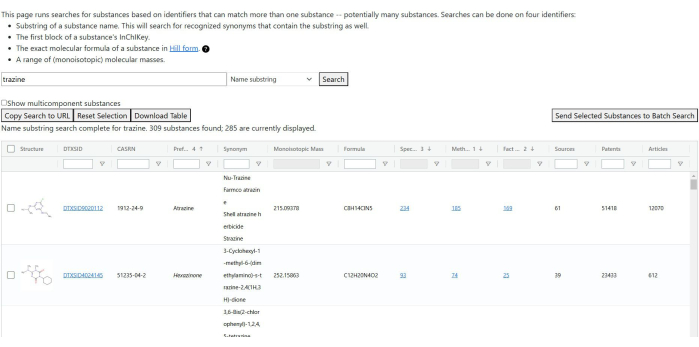
Abbildung 5: Suchergebnisse für Teilidentifikatoren für "trazine". Die Suche findet Substanzen mit bevorzugten Namen oder Synonymen, die den Teilstring "Trazine" enthalten. Zwei der drei Ergebnisse enthalten "Trazine" nur in ihren Synonymen, nicht in ihren bevorzugten Namen. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
3. Durchsuchen von Datensätzen
- Factsheet und Methodenlisten: Auf diesen Seiten werden alle Informationsblätter und Methoden aufgelistet, die sich in der Datenbank befinden, mit verschiedenen Möglichkeiten, sie zu filtern (siehe Abbildung 6). Da die Funktionalität der beiden Seiten weitestgehend gleich ist, werden sie hier gruppiert.
HINWEIS: Wenn Sie zur Seite navigieren, werden die Tabellen geladen. Dies kann aufgrund der Anzahl der vorhandenen Datensätze einen Moment dauern.- Nachdem eine Tabelle geladen wurde, verwenden Sie die Eingaben am oberen Rand jeder Spalte, um die Daten und verschiedene Felder zu filtern. Die genauen Felder variieren zwischen den Tabellen, aber die meisten können ausgewählt oder gefiltert werden.
- Verwenden Sie das Feld Vollständiger Tabellenfilter über der Tabelle, um alle Spalten auf eine bestimmte Zeichenfolge zu überprüfen.
HINWEIS: Die Methodenliste enthält zwei Felder, die standardmäßig ausgeblendet sind: Autor und Herausgeber. Der vollständige Tabellenfilter erfasst Datensätze, die den gesuchten Begriff in einem dieser Felder enthalten. - Die Merkblattliste ermöglicht es, einzelne Ergebnisse durch die Suche nach einem bestimmten Stoff zu filtern. Geben Sie einen Stoffnamen ein, CASRN, InChIKey oder DTXSID, und klicken Sie auf Suchen , um die Tabelle zu filtern. Klicken Sie auf Filter löschen , um den Substanzfilter zu löschen.
HINWEIS: In beiden Tabellen stehen die folgenden Schaltflächen zur Verfügung: Filter in Zwischenablage kopieren kopiert eine URL in die Zwischenablage, die beim Zugriff über einen Browser die Liste lädt und die Filterfelder in der Tabelle mit den aktuellen Werten vorausfüllt. Tabelle herunterladen lädt eine Liste aller sichtbaren Ergebnisse und Filter in der Tabelle herunter; Stoffe herunterladen lädt eine Liste aller Stoffe herunter, die in der (gefilterten) Tabelle erscheinen; Filter zurücksetzen löscht alle Tabellenfilter, einschließlich des vollständigen Tabellenfilters.
- Massenspektrumsuche: Führen Sie diese Suche durch, um eine Liste von Massenspektralübereinstimmungen aus der Datenbank abzurufen, die auf einem vom Benutzer bereitgestellten Spektrum basieren (siehe Abbildung 7).
- Füllen Sie die vier erforderlichen Eingabefelder aus oder passen Sie sie an: einen Massenbereich für die Zielsubstanz in Daltons, mit einer Fehlerspanne in Dalton oder Teilen pro Million (ppm); eine Methodik, entweder GC/MS oder LC/MS; ein Massenspektrum, das als Liste von Ladungs-Masse- und Intensitätspaaren angegeben ist; und die Größe des Massenfensters für die maximale Ähnlichkeit.
- Sobald diese Felder ausgefüllt sind, klicken Sie auf die Schaltfläche Suchen darunter.
HINWEIS: Wenn die Suche abgeschlossen ist und Spektren gefunden wurden, wird auf der rechten Seite der Seite eine Tabelle angezeigt, in der Spektren aufgelistet sind, die der ausgewählten Methodik aus allen Substanzen entsprechen, die dem Massenbereich entsprechen, sortiert nach der Entropieähnlichkeit zwischen dem vom Benutzer eingereichten Spektrum und dem Datenbankspektrum. - Wählen Sie eine Zeile in der Tabelle aus, um ein Diagramm anzuzeigen, das einen Vergleich des Benutzerspektrums mit dem Datenbankspektrum (jeweils am oberen und unteren Rand des Diagramms) anzeigt. Verwenden Sie das Feld Minimale Ähnlichkeit zum Anzeigen , um Ergebnisse auszublenden, die unter einer bestimmten Entropieähnlichkeit liegen.
- Visualisierung der Klassifizierung funktionaler Verwendungen: Diese Seite visualisiert die Ontologie der funktionalen Verwendung von AMOS und verlinkt zu den Methoden und Informationsblättern für diese Verwendungsklassen. Die Klassen werden in einem gerichteten Graphen dargestellt, wobei die Kanten von allgemeineren übergeordneten Klassen zu spezifischeren untergeordneten Klassen übergehen (siehe Abbildung 8).
- Verwenden Sie das Suchfeld auf der rechten Seite, um die Liste der funktionalen Verwendungsklassen zu durchsuchen. Bewegen Sie den Mauszeiger über den Namen einer Verwendungsklasse, um den entsprechenden Knoten im Diagramm hervorzuheben.
- Wenn Sie das Diagramm direkt untersuchen, zeigen Sie mit der Maus auf einen angegebenen Knoten, um eine kurze Beschreibung dieser Klasse anzuzeigen und alle direkten übergeordneten oder untergeordneten Klassen für diesen Knoten hervorzuheben.
- Klicken Sie mit der rechten Maustaste auf einen Klassennamen aus der Liste auf der rechten Seite der Seite oder auf einen Knoten im Diagramm, um ein Menü mit Optionen für die Methoden- und Informationsblattlisten aufzurufen. Wählen Sie eine dieser Optionen aus, und es wird eine neue Browserregisterkarte mit dieser Liste geöffnet, wobei das Feld für die Funktionsklasse mit der ausgewählten Funktionsklasse vorgefiltert ist.
- Ternäres Diagramm des Bodens: Diese Seite stellt die Klassifizierung der Bodentextur des US-Landwirtschaftsministeriums wieder her und ermöglicht die Suche nach den Methoden von AMOS nach Bodentyp.
- Zeigen Sie mit der Maus auf den Bereich des Diagramms, um Details zur Zusammensetzung anzuzeigen.
- Klicken Sie auf einen Bereich des Diagramms, um eine neue Registerkarte in der Methodenliste zu öffnen, in der das Matrixfeld nach der ausgewählten Bodenklassifizierung vorgefiltert ist.

Abbildung 6: Gefilterte Liste der Analysemethoden. Die Tabelle ist nach Analyt und Matrix gefiltert und zeigt nur Methoden, die mit PFAS (per- und polyfluorierte Alkylsubstanzen) in Wasser in Verbindung stehen. Die entsprechende Liste der Merkblätter ähnelt stark diesem Layout. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 7: Suchergebnisse für die Ähnlichkeit des Spektrums. Als Eingabe wird ein Koffeinspektrum aus der AMOS-Datenbank verwendet. Ähnliche Spektren werden nach Substanz gruppiert, mit einem maximalen Ähnlichkeitswert von 1,0. Das gespiegelte Diagramm zeigt das Eingabespektrum (oben) und ein ausgewähltes Datenbankspektrum (unten). Hellblaue Spitzen sind für die Eingabe eindeutig, orangefarbene Spitzen für die Datenbank stimmen überein, und dunkelblaue Spitzen werden gemeinsam genutzt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 8: Visualisierung der Klassifizierung der funktionalen Verwendung. Die hierarchische Struktur wird dargestellt, wenn der Cursor über den Knoten "Industriechemikalien" (gelb umrandet) bewegt wird. Die untergeordneten Klassen sind grün umrandet. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 9: Visualisierung des ternären Diagramms des Bodens. Das Diagramm zeigt Daten zur Zusammensetzung von Bodenproben. Eine QuickInfo oben rechts zeigt die genaue Zusammensetzung des Bereichs an, der sich derzeit unter dem Cursor befindet. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Ergebnisse
Die oben gezeigten Screenshots von AMOS zeigen typische Ergebnisse der einzelnen Suchen in der Anwendung, einschließlich der Suche nach interessanten Substanzen und zwischen Spektren, Datenblättern und Methoden. Die Vielfalt der Abfragemöglichkeiten in der Datenbank zielt darauf ab, die wahrscheinlichsten und nützlichsten Arten der Suche so abzudecken, dass eine tiefere Untersuchung der Daten und der Substanzen, auf die sie sich beziehen, möglich ist.
Um die Suche eines Benutzers zu erleichtern, ist ein Großteil der Funktionalität so miteinander verbunden, dass eine tiefere Untersuchung der verfügbaren Daten unterstützt werden soll. Als Beispiel für einen Arbeitsablauf verknüpft die Visualisierung der funktionalen Verwendungsklassifizierung mit Ansichten der Methoden und Informationsblätter, die sich auf diese Funktionsklasse beziehen, aus denen Stofflisten extrahiert und in die Chargensuche eingespeist werden können, oder es können einzelne Dokumente untersucht und einzelne Stoffe in diesen Dokumenten weiter untersucht werden. Da viele Substanzen in Methoden auch experimentelle Massenspektren in der Datenbank haben, kann ein Forscher schnell von einer Stoffkategorie zu einer Reihe von Methoden und Spektren übergehen, mit denen das Vorhandensein einer bestimmten Substanz getestet werden kann (siehe Abbildung 9).
Da die Ergebnisse stark davon abhängen, wonach gesucht wird und welche Suche bzw. Suchen ausgeführt werden, sind repräsentative Ergebnisse für die gesamte Anwendung schwer zu definieren. Insgesamt kann es zutreffender sein, einen "Erfolg" in Bezug auf die Benutzererfahrung zu beschreiben; In diesem Fall ist zu hoffen, dass im Allgemeinen Folgendes zutrifft: Dass die Methoden des Suchens und Filterns (und die Möglichkeit, zwischen verschiedenen Suchen und Filtern zu wechseln) effektiv sind, um zu identifizieren, welche Teilmengen von Informationen ein Benutzer wünscht; dass die Ergebnisse, die der Benutzer findet, genau und nützlich sind. Abbildung 10 zeigt einen Beispiel-Workflow, der AMOS-Funktionen veranschaulicht.

Abbildung 10: Beispiel-Workflow zur Veranschaulichung von AMOS-Funktionalitäten. Der Arbeitsablauf beginnt mit einer Klassifizierung der funktionellen Verwendung (Atemwegsmedikamente), filtert Methoden, die mit Atemwegsmedikamenten im Blut in Verbindung stehen, untersucht eine bestimmte Methode und identifiziert Spektren für eine Substanz, die in dieser Methode enthalten ist. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Diskussion
Während sich viele Projekte und Anwendungen auf das Sammeln und Standardisieren von Informationen aus einem einzigen Datensatztyp konzentrieren, wie z. B. Methoden, Datenblätter oder eine bestimmte Art von Spektren, ist AMOS das erste identifizierte Tool, das große Mengen an Informationen über mehrere Datensatztypen hinweg sammelt und integriert. Die Vereinheitlichung, Harmonisierung und Strukturierung von Daten aus diesen verschiedenen Quellen führt zu einer Datenbank, die leichter in Arbeitsabläufe integriert werden kann, die Zugang zu Methoden der analytischen Chemie erfordern. Die Möglichkeit, die Datenbank auf mehrere komplementäre Arten zu durchsuchen, ermöglicht ein effizientes Abrufen von Informationen, die andernfalls einen umfangreichen manuellen Aufwand über mehrere Websites oder Tools erfordern würden.
Vor der Veröffentlichung wurde der Nutzen von AMOS durch seine Verwendung durch EPA-Mitarbeiter zur Unterstützung einer Vielzahl von Projekten demonstriert. Die EPA hat ein anhaltendes Interesse an der Anwendung der Massenspektrometrie für die nicht-zielgerichtete Analyse10,11, und mehrere Initiativen haben die experimentellen Massenspektren in AMOS genutzt, um die Suche gegen eine große In-silico-Spektralbibliothek zu verbessern, die aus DSSTox-Chemikalien generiert wurde12,13. Andere Projekte haben die Suche nach struktureller Ähnlichkeit genutzt, um Ansatzpunkte für die Entwicklung neuer Methoden zu identifizieren, bestehende Methoden zur Bewertung von Nachweis- und Quantifizierungsgrenzen untersucht und Sammlungen von Chemikalien analysiert, die mit Methoden zur Bewertung des Ausmaßes der chemischen Raumabdeckung verknüpft sind.
Die Aggregation potenzieller Trainingsdaten durch AMOS unterstützt die Entwicklung quantitativer Modelle der Zugänglichkeit für analytische Methoden14, eine zentrale Anforderung für die Weiterentwicklung von Arbeitsabläufen für die nicht-zielgerichtete Analyse (NTA). Die Kurationsbemühungen innerhalb von AMOS erleichtern auch Initiativen zur Modellierung, Erforschung und Visualisierung chemischer Räume, die mit der methodischen Abdeckung verbunden sind14.
Während die Kernfunktionalität von AMOS ausgereift ist, wird die laufende Entwicklung durch das Feedback der Benutzer geleitet. Zu den aktuellen Aufgaben gehören die Einbindung zusätzlicher Daten, die Kuratierung weiterer Metadaten für eine verbesserte Filterung und die Erweiterung der Suchfunktionen. In Zusammenarbeit mit EPA-Stakeholdern werden Application Programming Interfaces (APIs) entwickelt, um den programmatischen Zugriff zu ermöglichen und Anwendungsfälle zu adressieren, in denen die grafische Benutzeroberfläche (GUI) ineffizient sein kann. Eine Seite mit Versionshinweisen wurde in die Anwendung integriert, um Code-Updates im Laufe der Zeit zu verfolgen und zu kommunizieren.
Neue Datensätze und Chemikalien kommen derzeit wöchentlich hinzu; Nach der öffentlichen Veröffentlichung wird jedoch mit einem langsameren Veröffentlichungsplan gerechnet. Obwohl erhebliche Anstrengungen unternommen werden, um die Genauigkeit der Datensätze und der zugehörigen Metadaten zu gewährleisten, stammen viele der Daten aus öffentlichen Datenbanken. Daher ist eine vollständige Überprüfung jedes Datensatzes nicht möglich, und die Benutzer sollten sich darüber im Klaren sein, dass die absolute Datengenauigkeit nicht garantiert werden kann.
Offenlegungen
Dieses Papier gibt nicht unbedingt die Ansichten oder Richtlinien der U.S. Environmental Protection Agency wieder.
Danksagungen
Die Autoren danken dem Kurationsteam für all seine Arbeit bei der Kuratierung von Chemikalien für die Datenbank sowie Joshua Powell, Asif Rashid und Freddie Valone für die technische Unterstützung beim Aufbau und Einsatz von AMOS. Wir danken auch Charles Lowe für seine Durchsicht des Manuskripts.
Materialien
| Name | Company | Catalog Number | Comments |
| Git | N/A | https://git-scm.com/ | Open-source version control system. |
| JavaScript | N/A | https://ecma-international.org/publications-and-standards/standards/ecma-262/ | Programming language. Defined by ECMA International standards. |
| PostgreSQL | PostgreSQL Global Development Group | https://postgresql.org/about/licence | Open-source database management system. |
| Python | Python Software Foundation | https://www.python.org/ | Open-source programming language. |
Referenzen
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten