Method Article
用于揭示放射敏感和放射抗性基因的全基因组 CRISPR 筛选
* 这些作者具有相同的贡献
摘要
通过应用全基因组 CRISPR/Cas9 筛选方法,给出了一种细致而结构化的方法来选择耐药和敏感的辐射基因。该协议还具有可能作为其他研究工作的多功能框架,以调查对临床给药的化学药物的耐药机制。
摘要
CRISPR-Cas9 系统已被利用并重新用于强大的基因组编辑工具。通过利用这项技术,研究人员可以精确地剪切、粘贴甚至重写活细胞内的 DNA 序列。然而,CRISPR 筛选技术的应用远远超出了单纯的实验。它是对抗遗传疾病的关键工具,系统地剖析复杂的遗传景观,使研究人员能够揭示生物现象背后的分子机制,并使科学家能够识别和定位癌症、囊性纤维化和镰状细胞性贫血等疾病的根本原因。其中,癌症对医学构成了巨大的挑战,刺激了根除工作。放疗作为一种传统治疗方法,效果好,但有局限性。它可以根除癌细胞,但也损害健康组织,造成降低生活质量的不利影响。此外,并非所有癌细胞都对放疗有反应,有些癌细胞可能会产生耐药性,使病情恶化。为了解决这个问题,引入了一种全面的全基因组 CRISPR 筛选技术,因为它能够有效识别放射敏感和放射抗性基因,从而推进癌症研究和治疗领域。按照所述方案对暴露于辐射的肺腺癌细胞进行全基因组 CRISPR 筛选,通过该方案鉴定了放射抗性和放射敏感性相关基因。
引言
对生物现象的研究本质上与对细胞行为的研究交织在一起,反过来,对细胞行为的检查从根本上与其基因组的探索有关。随着现代技术的不断发展,医学研究人员正逐渐将注意力转移到通过基因编辑改变细胞行为上,以提高各种疾病的治疗效果。在这方面,由于其应用相对简单,成簇规则间隔短回文重复序列 (CRISPR) 技术已成为基因组编辑的革命性工具1。CRISPR-Cas9 系统由 Cas9 核酸酶和单向导 RNA (sgRNA) 组成,可特异性识别并结合靶 DNA 序列,引导 Cas9 核酸酶在该位置切割,导致基因组 DNA 出现双链断裂 (DSB) 2,3,4。此外,其他物质的引入会导致基因组中的特异性插入、缺失或突变,从而实现靶向基因编辑。
在功能基因组学研究中,RNA 干扰 (RNAi) 筛选曾经是进行大规模功能丧失实验以研究基因在癌症中的作用的广泛使用的方法。RNAi 技术通过特异性沉默靶基因来研究基因功能,帮助研究人员识别关键的致癌因素。然而,它受到脱靶效应和不完全基因敲低效率的限制。脱靶效应可能导致其他非靶基因沉默,从而影响实验结果的准确性和可靠性 1,2。此外,RNAi 对某些基因的敲低效率较低,可能无法完全抑制靶基因表达。与传统的 RNAi 筛选相比,CRISPR 筛选表现出更高的特异性和效率3。该技术不仅可以精确编辑特定基因,还可以进行全基因组大规模筛选,为基因功能研究提供有力支持。CRISPR 筛选技术是一种基于 CRISPR-Cas9 系统的强大基因编辑工具,用于高效筛选和揭示细胞中特定基因的未知功能 5,6,7,8。研究人员针对特定基因或基因区域批量设计 sgRNA,并精确、严格地制备相应的 sgRNA 文库,确保其完整性和功能性9。然后将这些 sgRNA 文库封装到慢病毒颗粒中,用于有效感染宿主细胞。成功感染后,在个人定义的筛选条件下培养感染的细胞。筛选后,提取筛选细胞的基因组 DNA,保持高标准的纯度和数量。随后,对 sgRNA 目标的靶向区域进行 PCR 扩增,该过程可准确复制所需的核酸片段 3,9。最后,对扩增的 DNA 片段进行高通量测序,从而能够对目标区域进行全面高效的分析,从而为研究基因的功能和行为提供有价值的见解4。
癌症是一种复杂的疾病,对人类健康构成了巨大的威胁。在全球范围内,研究人员和临床医生正在齐心协力,以揭示致癌作用的分子机制并开发新的治疗策略。已经建立了国际合作,以加速基础研究结果向临床应用的转化,最终目标是改善患者的预后。Sasmal 等人提出了一种基于合成主客体系统的生物正交组装策略,用于精确靶向转移性癌细胞,这极大地帮助了数十名科学家推进医疗技术。他们出色的研究工作具有高度的创新和独特的见解,为科学界做出了有意义的贡献10.癌症的特征是基因组不稳定的动荡状态,这是由 DNA 损伤反应的不稳定调节引起的11-14。DNA 损伤包括单核苷酸缺陷、单链断裂和 DSB。同源重组 (HR) 和非同源末端连接 (NHEJ) 参与不同阶段的 DSB 修复 15,16,17。在此基础上,放疗已成为一种可行的治疗选择,它利用高能射线(如 X 射线和 γ 射线)照射肿瘤组织,在肿瘤细胞中造成 DNA 损伤,从而破坏其生长和增殖18。然而,放疗并不总是在很大一部分癌症患者中产生理想的效果,这可能是由于癌旁组织的损伤和肿瘤固有特征造成的限制,例如对放疗的低敏感性 19,20,21。
理论上,任何细胞类型都可用于 CRISPR 筛选。然而,在突变群体中保持足够的代表性需要大量的起始细胞。丰度低的细胞类型不是特别适合全基因组筛选。至于文库的选择,大多数文库每个靶基因包含 3-6 个 gRNA,维持每个 gRNA 在种群中的分布至关重要18。由于特异性 gRNA 的富集或耗竭而导致的代表性丧失可能导致结果分布不均匀。为了解决这个问题,选择经过市场测试的市售 CRISPR 文库可能是一个更可取的选择20。 体外 使用同质癌细胞系的 CRISPR 筛选可能无法完全捕获 体内 肿瘤的遗传和表观遗传异质性。虽然 体外 筛选揭示了参与 DNA 损伤修复和辐射诱导的自分泌信号传导的关键基因,但它并没有完全复制肿瘤微环境,包括缺氧诱导的放射抗性(通过 ROS、代谢适应和自噬)、免疫介导的旁分泌效应和 ECM 依赖性细胞因子调节。在使用 CRISPR 筛选探索与辐射敏感性或耐药性相关的基因之前,必须仔细考虑这些因素。鉴于目前的治疗形势,迫切需要确定并深入研究与放射耐药性和放射敏感性相关的因素,以有效提高放射治疗效果22。鉴于 CRISPR 筛选在研究未知基因功能方面的关键优势,提供了一种系统详细的全基因组 CRISPR 筛选技术,以有效识别放射敏感和放射抗性基因。
研究方案
本研究中使用的试剂和设备列在 材料表中。
1. 选择合适的辐射剂量
- 贴壁细胞的制备和铺板
- 使用含有 10% FBS 的 RPMI 1640 完全细胞培养基,将细胞密度调节至 5 x 105 个细胞/mL,用于传代和细胞生长。将细胞分配到 3.5 cm 培养皿中,以不同剂量进行辐射。向每个培养皿中加入 2 mL(1 x 106 个细胞),并在 37 °C 和 5% CO2 下孵育过夜。
- 应用不同的辐射剂量
- 将 3.5 厘米的培养皿从 1 到 5 编号。使用组 #1 作为对照组,其余 4 组指定为治疗组。
- 用密封膜密封 6 孔板的边缘,用于治疗组,并分别施用 2、4、6 和 8 Gy 的辐射剂量。处理后,将培养皿放回培养箱中。
- 放疗后细胞系的铺板
- 准备 6 孔板和 96 孔板。用 PBS 洗涤 3.5 cm 培养皿中的细胞一次,用 0.25% 胰蛋白酶消化对数生长的细胞,并使用含有 10% FBS 的 RPMI 1640 完全培养基将细胞密度调节至 1 x 105 个细胞/mL。
- 将 10 μL/孔(1,000 个细胞/100 μL)接种到 6 孔板中,每个辐射剂量重复 3 次。向每个孔中加入 2 mL 完全培养基,并在 14 天后计数(当每个克隆组有大约 50 个细胞时)。
- 将 30 μL/孔(3,000 个细胞/100 μL)接种到 96 孔板中,每个辐射剂量重复 5 次。向每个孔中加入 70 μL 完全培养基,并在 72 小时后测量细胞活力。
- 细胞活力测量
注:克隆形成抑制率 = 1 - (处理组中的克隆数/对照组中的克隆数)x 100%。CCK-8 测定利用水溶性四唑盐 (WST-8),该盐可被细胞脱氢酶还原,生成高度水溶性黄色甲臜产物11。甲臜的产生量与活细胞的数量成正比,其光密度(在 450 nm 波长下测量)准确反映了细胞代谢活性和增殖状态11,13。基于这一公认的原理,CCK-8 检测已被广泛用于各种应用,包括细胞增殖检测和肿瘤药物敏感性检测。在该方案中,采用 CCK-8 测定法系统评估不同辐射剂量下的细胞活力。- 将 CCK8 试剂与不含 FBS 的 RPMI 1640 培养基以 1:9 的比例混合。丢弃 96 孔板中的培养基,并向每个孔中加入 100 μL 含 CCK8 的培养基。
- 在黑暗中孵育 1 小时,并使用酶标仪测量 450 nm 处的 OD 值。
注:细胞存活率 = [(处理组的 OD 值 - 空白孔的 OD 值) / (对照组的 OD 值 - 空白孔的 OD 值)] x 100%
- 辐射剂量的选择
- 将克隆形成抑制率与细胞存活率相结合,以便根据研究需要选择合适的辐射剂量。选择抑制率为 50% 的辐射剂量,用于筛选放射抗性和放射敏感基因。
2. 选择合适的 MOI 和嘌呤霉素浓度
- 贴壁细胞的铺板
- 将细胞密度调节至 3 x 105 个细胞/mL,并将每孔 1 mL(3 x 105 个细胞)接种到 12 孔板中。将板在 37 °C 和 5% CO2 下孵育过夜。
- 慢病毒感染
- 使用移液器从 12 孔板中吸出培养基,并用 1 mL 含有 10% FBS 的 RPMI 1640 完全培养基替换。准备经过质量检查的全基因组 CRISPR 慢病毒文库(建议使用市售文库)并设置对数浓度梯度(例如,0-10-50-100-200-400-800)4,5,6,7,8。
- 将相应量的慢病毒添加到 2 μL/皿的聚凝胺中,并在室温下平衡 5 分钟。将慢病毒和聚凝胺混合物缓慢滴入每个孔中,充分混合,并在 37 °C 和 5% CO2 下孵育过夜。
- 测定用于细胞杀伤的最低嘌呤霉素浓度
- 将亲代细胞密度调节至 3 x 105 个细胞/mL,并将每孔 1 mL(3 x 105 个细胞)接种到 12 孔板中。将板在 37 °C 和 5% CO2 下孵育过夜。
- 将嘌呤霉素以 0-0.1-0.2-0.5-1-2-4-8 μM 的浓度梯度添加到 12 孔板的每个孔中。72 小时后计数细胞。杀死孔中所有细胞的最低浓度是用于细胞杀伤的最低嘌呤霉素浓度,将用于后续病毒感染细胞的筛选。
- 感染后嘌呤霉素的选择
- 感染后的第二天,从每个孔中吸出培养基,并用 1 mL 含有 10% FBS 的 RPMI 1640 完全培养基替换。继续培养 48 小时。
- 感染 72 小时后,用含有用于细胞杀伤的最低嘌呤霉素浓度的完全培养基替换现有培养基。在 12 孔板中设置 2 个孔,没有慢病毒感染作为阴性和阳性对照。用嘌呤霉素处理阴性对照,不处理阳性对照。继续培养 72 小时。
- 嘌呤霉素选择 72 小时后,阴性对照中的所有亲代细胞都将死亡,而阳性对照中的亲本细胞则显示出最小的死亡。计算每个孔的 MOI。
注:MOI =(病毒感染组中的细胞数/阳性对照组中的细胞数)x 100%。使用 MOI 为 ~0.3 的病毒浓度进行后续筛选。
3. 全基因组 CRISPR 慢病毒文库感染
- 贴壁细胞系的接种
- 在含有 10% FBS 的 RPMI 1640 完全培养基中,将细胞密度调节至每毫升 1 x 107 个细胞,用于传代和细胞生长。将 1 mL 细胞(1 x 107 个细胞)接种到每个 15 cm 培养皿中,在 37 °C 和 5% CO2 下接种 8 小时。一旦细胞粘附并达到 70%-80% 的汇合度,它们就准备好进行病毒感染(拷贝数约为 500)。
- 慢病毒感染
- 从 15 cm 培养皿中吸出培养基,并用 15 mL 含有 10% FBS 的 RPMI 1640 完全培养基替换。准备经过质量检查的全基因组 CRISPR 慢病毒文库。
- 加入相应量的 MOI = 0.3 至 30 μL/皿聚凝胺慢病毒,并在室温下平衡 5 分钟,将慢病毒和聚凝胺混合物缓慢滴入 15 cm 培养皿中,充分混合,并在 37 °C 下用 5% CO2 孵育过夜。同时,准备一个 15 cm 的培养皿,以亲本细胞作为对比。
- 感染后嘌呤霉素的选择
- 在病毒感染后的第二天,从 15 cm 培养皿中吸出培养基,并用 15 mL 含有 10% FBS 的 RPMI 1640 完全培养基替换。继续培养 48 小时。
- 感染后 72 小时,用含有最低致死浓度嘌呤霉素的完全培养基替换培养基。以相同的方式将未感染的亲本细胞视为阴性对照,并继续培养 72 小时。嘌呤霉素选择 72 小时后,阴性对照组中的亲本细胞将被全部杀死,慢病毒感染中存活的细胞被认为成功感染。
- 提取第 0 天的基因组
- 使用 0.25% 胰蛋白酶从一个 15 cm 培养皿中消化细胞,重悬于含有 10% FBS 的 RPMI 1640 完全培养基中,并计数细胞数量。将此样本标记为第 0 天。
- 以 300 x g 离心 5 分钟(在室温下)并弃去上清液。重悬于 1 mL PBS 中,以 300 x g 离心 5 分钟,然后弃去上清液。提取第 0 天基因组 DNA,使用 nanodrop 紫外分光光度计测量 DNA 浓度和纯度。
- 对于 sgRNA 完整性检测,使用琼脂糖凝胶电泳分析扩增的 sgRNA 文库,确保条带清晰且无明显降解,从而保持文库的完整性23。对于慢病毒文库覆盖率评估,采用深度测序技术对文库内的 sgRNA 进行测序分析24,25。
- 在 CRISPR 筛选中,PCR 扩增可作为 sgRNA 片段完整性和文库质量的初步验证6。为了更全面地分析文库覆盖率和 sgRNA 分布模式,并确保在筛选过程中每个靶基因的 sgRNA 充分代表性,请进行 NGS 以深入了解筛选9 的稳健性。
注:通过检测筛选前后 sgRNA 丰度的变化,并识别潜在的 sgRNA 脱靶整合或文库污染,可以进一步提高 CRISPR 筛选的准确性和可靠性。这些第 0 天样品作为关键的阴性对照,并通过 PCR 扩增和 NGS 进行全面质量评估,以实现 2 个关键目标:(1) 确认必需基因耗竭(表明文库充分表达),以及 (2) 证明非必需基因中稳定表达(建立实验基线条件)9。 - 确保覆盖率达到预期水平,确保文库的多样性和代表性,避免筛选过程中的偏差。对于感染效率测量,使用荧光成像评估感染效率,确保其满足实验要求。
4. 应用辐射作为筛查条件
- 分组
- 将 CRISPR 感染细胞的密度调节至每毫升 1 x 107 个细胞,并将 1 mL 接种到每个 15 厘米培养皿中。在 37 °C 下用 5% CO2 孵育过夜。
- 放射治疗
- 将细胞随机分为 2 组:处理组和对照组,每组 6 个培养皿。对治疗组的细胞进行适当剂量的放射治疗,而让对照组的细胞不经处理以正常繁殖。照射后,继续在 37 °C 和 5% CO2 下孵育 7 天。
- 在第二周,重复对治疗组细胞进行适当剂量的辐射,让对照组细胞不处理并让它们正常繁殖。放疗后,继续在 37 °C 和 5% CO2 下再孵育 7 天。
5. 基因组提取和测序
- 第 14 天的提取
- 处理 14 天后,用 0.25% 胰蛋白酶消化处理组和对照组的细胞,使用含有 10% FBS 的完全 RPMI 1640 培养基重悬它们,并将它们标记为第 14 天-RT 或第 14 天-NC。
- 将细胞以 300 x g 离心 5 分钟,然后弃去上清液。重悬于 1 mL PBS 中,以 300 x g 离心 5 分钟,然后再次弃去上清液。提取第 14 天的基因组 DNA,并使用 nanodrop UV 吸光度光谱检测 DNA 的浓度和纯度。
- PCR 扩增
- 准备序列的相应引物(参见 材料表)并将它们稀释至 10 μM.将试剂添加到无菌微量离心管中,建立 20 μL 反应系统,以 300 x g 离心 5 秒,并充分混合。根据扩增条件设置 PCR 仪器参数,得到 PCR 产物。
- 琼脂糖凝胶电泳检测
- 准备凝胶铸板,用琼脂糖密封模具边缘,插入梳子,根据样品 DNA 的长度制备适当浓度的琼脂糖凝胶。
- 精密称取一定量的琼脂糖粉,加入适量电泳缓冲液,混匀,置微波炉中加热熔化。稍微冷却后,加入适量的核酸染料,轻轻混匀,缓缓倒入凝胶流延模具中。让凝胶凝固 30 分钟。
- 取下电泳梳,向电泳槽中加入适量的电泳缓冲液,直至其覆盖凝胶。向 DNA 样品中加入适量的上样缓冲液,充分混匀,然后用移液管将混合物缓慢加入样品孔中。
- 根据 DNA 片段的大小和琼脂糖凝胶的浓度设置适当的电压。电泳后,小心取出凝胶并将其放入凝胶成像仪中,观察结果并检查 PCR 扩增是否成功。
- Illumina 测序
- 从三组(Day 0 组、对照组和治疗组)收集基因组 DNA,并将其发送给一家公司进行文库构建和 Illumina 测序。对测序结果进行生物信息学分析和可视化,以获得放疗敏感和放疗耐药的基因26。
- 设置多个重复实验进行数据分析,并对实验结果进行统计分析,确保数据的一致性和重现性27,28。
- 测序数据的质量评估
- 对原始数据进行严格的过滤和质量控制机制,以保证测序信息的精度5.使用指定的转换算法和评估碱基检出期间发生错误可能性的模型,根据测序错误率计算每个核苷酸的 Phred 分数 (Qphred)28。
- 将每个碱基位置的测序错误率保持在 1% 以下(相当于 Q30 阈值),并确保至少 80% 的测序数据达到该 Q30 标准,以支持后续分析程序。
- 数据处理和样品级质量控制
注:有关详细信息,请参阅以前发布的报告29,30。- 将测序数据中过滤的读数与 sgRNA 文库序列进行比对。报告统计数据,包括文库中完美比对的 sgRNA 数量、sgRNA 的平均丰度、未检测到的 sgRNA 数量以及成功映射到 sgRNA 文库的单个样品的读数比例。使用较高的映射比率表示覆盖范围更大。
- 计算每个样品中每个基因(靶向不同 sgRNA)的读数富集。使用 MAGeCK 的“中位”归一化方法对样品中每个 sgRNA 的支持读数进行归一化。通过评估 sgRNA 读取计数的分布、生成箱线图、进行主成分分析 (PCA) 和构建相关热图来执行样品级质量控制。
- 假设 sgRNA 读取计数服从泊松分布。在箱形图中描述来自不同组的标准化 sgRNA 计数,以可视化样品内的整体数据分布并比较组间分布。
- 使用 PCA 通过反映主成分之间的差异和每个成分内的变异率来简化数据分析,从而促进对组内和组间变异的观察。使用相关性热图来说明样本之间的关系。
- 基本分析
- 进行组间差异 sgRNA 分析,并在质量控制和初步数据处理后确定必需基因。对相关基因进行功能富集分析30.根据 MAGeCK 或其他统计方法(如 MLE)计算的稳健秩聚合 (RRA) 分数对基本基因进行排名,RRA 分数越低表示重要性越高。
- 使用 R 语言或其他编程语言对排名结果进行生物信息学分析,并使用 GO 和 KEGG 分析图30 可视化结果。通过 ID 映射或序列注释将基因或蛋白质与相应的 GO 术语(分子功能、生物过程和细胞成分)联系起来。
- 使用 KEGG 作为数据库来了解高级功能和生物系统,将已识别的基因目录与细胞、物种和生态系统水平的系统功能联系起来。从 GO 和 KEGG 分析中选择前 10 或 20 条通路,以直观地显示通路方向。
结果
肺癌的死亡率最高,是一种高度侵袭性和普遍的医学疾病。以肺癌细胞系 A549 为例,以辐射为筛选条件进行全基因组 CRISPR 筛选,示意图工作流程如图 1 所示。首先,通过克隆形成和 CCK8 实验探索 A549 细胞对不同剂量辐射的敏感性(图 2)。在克隆形成测定中,2 Gy 时菌落计数为 140 ± 5.35,而 0 Gy 时为 303 ± 31.63,而在 CCK-8 测定中,2 Gy 时光密度 (OD) 值为 0.65 ± 0.05,而 0 Gy 时为 1.35 ± 0.08。要研究放射敏感性和放射抗性候选基因,请选择 2 Gy (IC50) 作为后续辐射剂量。用不同剂量的嘌呤霉素处理 A549 细胞,并在 72 小时后计数。检测到 A549 细胞的嘌呤霉素的最低杀伤浓度为 1 μM(图 3)。A549 细胞用不同梯度 MOI 的慢病毒感染。感染 72 h 后,荧光显微镜观察感染效率,并在嘌呤霉素处理 72 h 前后计数。获得了感染效率为 ~0.3 的 MOI(表 1)。人类基因组包含 19,050 个基因,每个基因对应 6 个 sgRNA,拷贝数为 500(19,050 x 6 x 500 = 5.7 x 107 个细胞)。使用 MOI = 0.3 的慢病毒,因此感染 5.7 x 107 个 A549 细胞。72 小时后,用 1 μM 嘌呤霉素再处理细胞 72 小时。收获第 0 天的基因组,并进行 PCR 扩增。CRISPR 全基因组文库中的 sgRNA 序列显示 231 个碱基对的长度,琼脂糖凝胶电泳结果证实了这一点(图 4)。
PCR 产物随后被送往 Novogene 进行数据质量控制和样品级质量控制。映射比率产生了大约 60% 的综合覆盖率,考虑到整个基因组处理过程中的绝对复杂性和不可避免的损耗,这个指标被认为足以用于全基因组筛选程序(图 5)。sgRNA 读取计数遵循泊松分布,符合理论预期。随后通过 PCA 绘图和相关热图的分析生动地描绘了不同组之间的可识别差异,组间差异明显大于组内差异。此外,分组样本内的变化率在可接受的范围内,证实了样本水平质量控制措施的成功。随后,将利用 MAGeCK 设计的 RRA 排名,利用 R 语言对排名结果进行基础生物信息学评估。值得注意的是,GO 术语中的前 15 条通路突出了与 DNA 损伤相关的机制,这与实验的基本筛选标准无缝一致。
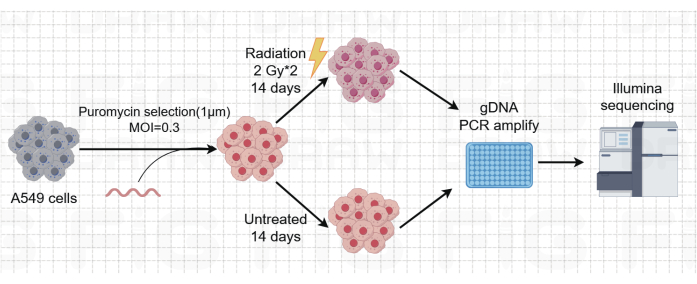
图 1:A549 肺癌细胞中全基因组放疗 CRISPR/Cas9 筛选示意图。 使用全基因组 CRISPR/Cas9 和放疗在 A549 细胞中筛选工作流程的示意图。 请单击此处查看此图的较大版本。
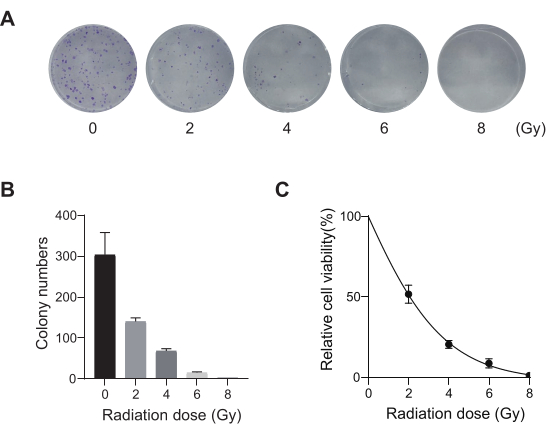
图 2:A549 细胞的辐射剂量优化。 (A) A549 细胞暴露于增加剂量的辐射(0 Gy、2 Gy、4 Gy、6 Gy 和 8 Gy)并使其生长 14 天。显示了菌落形成测定图像。结果代表了两个独立的生物实验。(B) 克隆形成分析图像的定量结果。误差线表示标准差 (n = 3)。单因素方差分析得出 P < 0.01。结果代表了两个独立的生物实验。(C) A549 细胞暴露于不同辐射剂量后的剂量反应生存曲线。误差线表示标准差 (n = 3)。单因素方差分析得出 P < 0.01。结果代表了两个独立的生物实验。 请单击此处查看此图的较大版本。
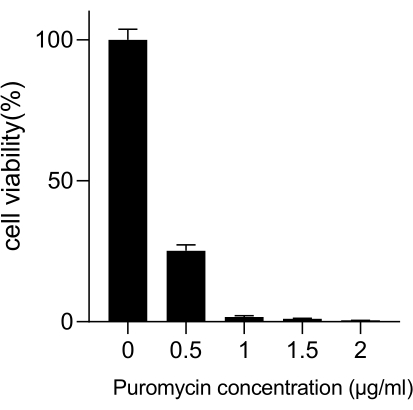
图 3:测定 A549 细胞筛选所需的最低嘌呤霉素浓度。 用浓度递增的嘌呤霉素处理 A549 细胞并孵育 72 小时。误差线表示标准差 (n = 3)。单因素方差分析得出 P < 0.01。结果代表了两个独立的生物实验。 请单击此处查看此图的较大版本。

图 4:通过琼脂糖凝胶电泳验证 PCR 产物。 琼脂糖凝胶电泳用于检测 PCR 产物条带的存在和质量。 请单击此处查看此图的较大版本。
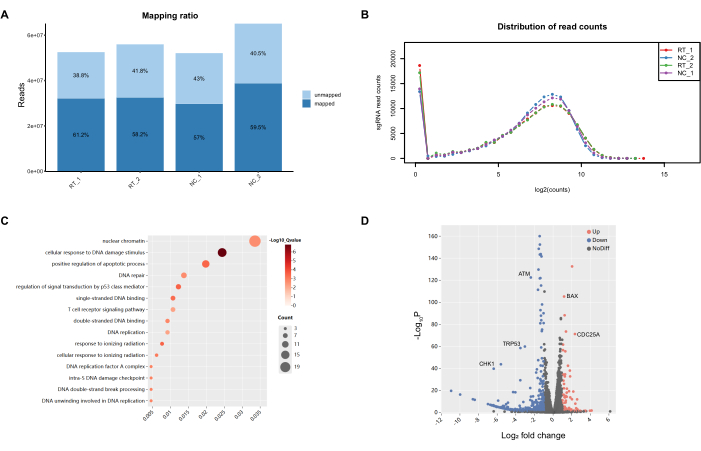
图 5:PCR 扩增产品的质量控制和基本分析。 (A) 作图比率表明参考基因组的覆盖率约为 60%。(B) sgRNA 的读取计数分布服从泊松分布。(C) 前 10 个基因本体论 (GO) 术语中的大多数与 DNA 损伤反应相关。(D) 显示基因富集模式的火山图,红色为负富集基因,蓝色为正富集基因,灰色为无显著基因。 请单击此处查看此图的较大版本。
| 病毒颗粒 数字 (VP) | 计数编号 (嘌呤霉素治疗前) | 计数编号 (嘌呤霉素治疗后) | 感染 效率 |
| 1.23 × 104 | 1.23 × 105 | 1.04 × 104 | 0.084552846 |
| 3.72 × 104 | 1.24 × 106 | 3.61 × 104 | 0.291129032 |
| 6.15 × 104 | 1.23 × 107 | 5.4 × 104 | 0.43902439 |
表 1:不同病毒颗粒下嘌呤霉素选择前后的 A549 细胞计数。 在不同病毒颗粒下嘌呤霉素处理之前和之后记录的 A549 细胞计数。
讨论
CRISPR 筛选作为一项前沿的基因编辑技术,在科研领域引发了深刻的变化5.该技术起源于 CRISPR-Cas9 系统,由于其高效和精确性,已成为研究基因功能的重要工具9。CRISPR/Cas9 工程原理包括设计和引入具有大约 20 个核苷酸的特异性 sgRNA,以指导 Cas9 核酸酶精确定位和切割靶标 DNA 序列,从而实现基因编辑,如敲除、激活或抑制6。这一原则是高通量和可定制的 CRISPR 筛选开发的基础。通过构建包含大量 sgRNA 的文库,研究人员可以同时编辑和筛选多个基因,快速识别与特定表型或疾病状态相关的基因。此外,CRISPR 筛选允许在各种细胞类型、组织甚至整个生物体中进行实验,从而更全面地了解基因功能和作用机制。
随着技术的加速发展,CRISPR 筛选在各个领域都显示出巨大的应用潜力。例如,CRISPR 筛选可以帮助科学家发现新的药物靶点,从而加速药物发现和开发的过程。在疾病研究中,该技术揭示了疾病发病机制,为诊断和治疗提供了新的见解和方法。CRISPR 筛选在几个关键方面与传统筛选方法(如 RNAi 和小分子抑制剂)有根本区别:从机制上讲,CRISPR 通过基因编辑实现永久性基因修饰,而 RNAi 仅暂时抑制基因表达,小分子抑制剂可逆地阻断蛋白质功能28,29.在特异性方面,CRISPR 通过准确的 DNA 靶向表现出卓越的精度,而 RNAi 容易出现脱靶效应 3,5。关于效果持续时间,与传统方法的瞬时干预相比,CRISPR 会产生永久性的基因改变。此外,CRISPR 可实现全基因组筛选能力,可以同时研究多个基因/通路,同时在不同细胞类型中保持高效率9。传统方法通常仅限于特异性通路分析,并且通常在某些细胞系中疗效降低3。这些独特的特性使 CRISPR 筛选在功能基因组学研究和靶标发现方面特别有价值,尤其是在需要长期观察或精确遗传调控的研究中,它显示出优于传统筛选方法的独特优势。
然而,这个过程的特异性并不是绝对的,Cas9 蛋白可能会错误地识别和切割与预期靶序列相似但不完全相同的 DNA 区域5。这种情况通常称为脱靶效应。这种意外的 DNA 切割会导致基因组不稳定和不可预测的遗传后果,尤其是在人类治疗中应用时。CRISPR 技术在处理复杂的基因组编辑任务时也很困难。当面临包含众多基因、基因簇或复杂遗传设置的编辑需求时,CRISPR 系统在精确预测和指导编辑过程的结果方面可能会遇到限制1。
使用 CRISPR 技术进行全基因组筛选的主要技术障碍源于基因组的巨大复杂性,其中包含数万个基因,从而显著增加了脱靶效应的风险9。为了确保后续测序结果的完整性和可靠性,必须用单个 sgRNA23 转染每个细胞。这需要实验者对 MOI 进行细致的控制,确保其保持在 0.5 以下,同时保持足够的拷贝数阈值,该阈值必须至少为 500 或更高24。鉴于整个基因组中数以万计的基因中的每一个都需要使用 4-6 个 sgRNA,因此整个筛选过程需要大量的细胞25。实验持续时间延长、涉及的细胞数量庞大以及风险升高,因此实验者需要制定策略来缓解这些问题26。需要用 RNA-seq 以及功能增益和丧失测定进一步验证潜在基因。在接下来的实验中,在使用 CRISPR 编辑获得已鉴定基因的过表达和敲除细胞后,进行全基因组基因组学和转录组学以获得潜在的脱靶改变,特别是在可能经历非特异性 Cas9 切割的具有部分 sgRNA 互补性的基因座。COSMID 等生物信息学预测工具可能有助于识别高风险脱靶位点。拯救测定也可用于确保已鉴定基因参与的特异性。
在随后的分析中,差异表达 sgRNA 的标准依赖于 MAGeCK 计算的稳健秩聚合 (RRA) 评分,其中涉及统计显着性、倍数变化和重复一致性31,32。对于数据集,每个基因都根据结合基因表达水平和统计显著性的综合标准进行排名。RRA 利用 rank 的 Spearman 秩相关系数来评估所有数据集中每个基因的综合秩情况,并整合多个基因表达数据集的排序结果。在大多数数据集中排名相对较高的基因可能被视为真正的差异表达基因33。RRA 小于 0.05 的 p 值被认为具有统计学意义。至少 1.5 倍增加或减少 33% 的倍数变化被视为差异表达。此外,需要将 3 个独立重复的一致性视为统计学上不同的表达。在必需基因的 RRA 排名之后,较低的 RRA 分数表示较高的重要性。
作为基因组编辑的有力工具,CRISPR-Cas9 技术在研究辐射抗性区域方面表现出无与伦比的潜力和准确性26,27。但是,它的应用程序仍然存在局限性。除了脱靶效应的风险外,与放射敏感性相关的复杂性还给筛选过程带来了复杂性27。这种敏感性不仅受单个基因的控制,而且是多个基因及其调控网络之间错综复杂的相互作用的结果26。因此,尽管成功鉴定了与放射敏感性有关的特定基因,但它们可能只代表了更广泛情况的一小部分,对全面阐明潜在机制构成了重大挑战 34,35,36。以下是 CRISPR 筛选中常见问题的解决方案,例如感染效率低、sgRNA 降解和测序失败:由于 CRISPR 筛选需要 MOI 值 <0.5,因此细胞状态调整对于确保细胞处于对数生长期或通过荧光测定评估感染效率至关重要;sgRNA 降解的潜在原因包括核酸酶污染或载体设计缺陷,因此应避免 sgRNA 文库的重复冻融循环(在 -80 °C 下储存等分试样),并应使用 NGS 测序来验证 sgRNA 文库的完整性;测序失败可能是由于测序深度不足或 sgRNA 引物设计问题造成的,建议使用双端测序以提高 sgRNA 序列鉴定的准确性,建议测序深度为 >500×覆盖度37、38、39、40、41、42。
放疗仍然是癌症治疗的基石,但在不同肿瘤类型甚至相同癌症患者中的治疗效果也表现出显着的差异9。这种个体间异质性通常需要联合疗法来实现最佳肿瘤根除11,12。在临床实践中,放疗与免疫疗法(特别是免疫检查点抑制剂和抗体-药物偶联物)的整合已成为一种突出的治疗范式19。该方案提出了一种使用相对同质的癌细胞系的体外 CRISPR 筛选,它不能完全反映体内肿瘤的遗传和表观遗传异质性。在确定适合筛选的辐射剂量后,结果提供了潜在的放射敏感或放射抗性基因,这些基因需要在体外细胞测定和体内动物模型中进一步确认。此外,体内 CRISPR 筛选加单细胞转录组学可能有助于破译肿瘤异质性的复杂性。体外 CRISPR 筛选确定了细胞内 DNA 损伤修复的重要基因,以及响应辐射的自分泌机制,但实际上并没有完全复制 TME 异质性。例如,缺氧会增加 ROS 的产生,触发一个反馈回路,刺激代谢适应、抗氧化剂产生和自噬激活,最终促进放射抗性18。辐射后,免疫细胞可能在旁分泌机制中被激活,从而诱导放射敏感性14。ECM 是生长因子的储存库,细胞因子对放射敏感性有积极和消极的影响21。应进行体内 CRISPR 筛选以反映 TME 的异质性。此外,体外 CRISPR 筛选只能识别细胞内 DNA 损伤修复的重要基因,以及响应辐射的自分泌机制中的重要基因,而不能完全捕捉体内对放射敏感性的影响,包括肿瘤脉管系统和全身效应20,21。应进行体内 CRISPR 筛选以确定涉及体内放射敏感性的潜在因素。
在这里,设计了一个旨在利用 CRISPR 全基因组筛选的综合方案来查明与放疗的敏感性或耐药性有关的基因。通过揭示新的途径,该方法旨在为肿瘤放射治疗研究的进步铺平道路。该方案也可作为探索化疗临床限制机制和替代治疗药物进展的参考。总之,CRISPR 技术具有巨大的潜力和价值,但其固有的限制强调了清醒认识和明智方法的重要性。随着对未来研究和应用的深入研究,必须不懈努力探索和优化 CRISPR 技术,以克服其限制并充分发挥其潜力。
披露声明
没有。
致谢
本研究得到了湖北省区域科技创新项目 (2024EIA001) 和武汉大学中南医院医学科技创新平台建设支撑项目 (PTXM2025001) 的支持。 图 1 是使用 Figdraw 创建的。
材料
| Name | Company | Catalog Number | Comments |
| 150 mm cell and tissue culture dish | Wuxi NEST Biotechnology Co., Ltd. | 715011 | Cell culture |
| 35 mm cell and tissue culture dish | Wuxi NEST Biotechnology Co., Ltd. | 706011 | Cell culture |
| A549 cell line | ATCC | - | - |
| Cell counting kit-8 | Shanghai Beyotime Biotech Inc | C0041 | For cell viability assay |
| CO2-independent medium | PHCbi | MCO-50AIC | Cell culture |
| Countess 3 FL automated cell counter | Themo Scientific | AMQAF2001 | For cell counting |
| EDTA | Gibco | 25200056 | Cell culture |
| Fetal bovine serum | Gibco | 10099141 | Cell culture |
| Fluorescence microscopy | LEICA | ebq 100-04 | For fluorescence microscope |
| Genome-wide CRISPR lentiviral library | Shanghai OBiO Technology Co., Ltd. | H5070 | For lentiviral infection |
| MiniAmp plus PCR | Themo Scientific | C37835 | For PCR amplification |
| NEST cell culture plates, 12-well | Wuxi NEST Biotechnology Co., Ltd. | 712002 | Cell culture |
| NEST cell culture plates, 6-well | Wuxi NEST Biotechnology Co., Ltd. | 703002 | Cell culture |
| NEST cell culture plates, 96-well | Wuxi NEST Biotechnology Co., Ltd. | 701001 | Cell culture |
| PBS | Gibco | C10010500BT | Cell culture |
| PCR forward primer (AATGGACTATCATATGCTTACCGTAACTTGAAAGTATTTCG) | Beijing AuGCT Biotech Co., Ltd. | - | For PCR amplification |
| PCR reverse primer (GATGTGCGCTCTGCCCACTGACGGGCA) | Beijing AuGCT Biotech Co., Ltd. | - | For PCR amplification |
| Polybrene | Shanghai Beyotime Biotech Inc | C0351 | For lentiviral infection |
| Puromycin | MCE | HY-K1057 | For seletion post lentiviral infection |
| RPMI 1640 cell culture medium | Gibco | 23400-021 | Cell culture |
| TIANGENamp genomic DNA kit | Beijing TIANGEN Biotech Co.,Ltd. | DP304 | For genomic DNA extraction |
| X-ray powder diffractometer | PerkinElmer | For radiotherapy |
参考文献
- Barrangou, R., Marraffini, L. A. CRISPR-Cas systems: Prokaryotes upgrade to adaptive immunity. Mol Cell. 54 (2), 234-244 (2014).
- Joung, J., et al. Genome-scale CRISPR-Cas9 knockout and transcriptional activation screening. Nat Protoc. 12 (4), 828-863 (2017).
- Sanjana, N. E., Shalem, O., Zhang, F. Improved vectors and genome-wide libraries for CRISPR screening. Nat Methods. 11 (8), 783-784 (2014).
- Arroyo, J. D., et al. A Genome-wide CRISPR death screen identifies genes essential for oxidative phosphorylation. Cell Metab. 24 (6), 875-885 (2016).
- Sidik, S. M., et al. A genome-wide CRISPR screen in toxoplasma identifies essential apicomplexan genes. Cell. 166 (6), 1423-1435 (2016).
- Jing, Y., et al. Genome-wide CRISPR activation screening in senescent cells reveals SOX5 as a driver and therapeutic target of rejuvenation. Cell Stem Cell. 30 (11), 1452-1471 (2023).
- Shifrut, E., et al. Genome-wide CRISPR screens in primary human t cells reveal key regulators of immune function. Cell. 175 (7), 1958-1971 (2018).
- Shalem, O., et al. Genome-scale CRISPR-Cas9 knockout screening in human cells. Science. 343 (6166), 84-87 (2014).
- Katti, A., Diaz, B. J., Caragine, C. M., Sanjana, N. E., Dow, L. E. CRISPR in cancer biology and therapy. Nat Rev Cancer. 22 (5), 259-279 (2022).
- Sasmal, R., et al. Synthetic host-guest assembly in cells and tissues: Fast, stable, and selective bioorthogonal imaging via molecular recognition. Anal Chem. 90 (19), 11305-11314 (2018).
- Roos, W. P., Thomas, A. D., Kaina, B. DNA damage and the balance between survival and death in cancer biology. Nat Rev Cancer. 16 (1), 20-33 (2016).
- Huang, R., Zhou, P. K. DNA damage repair: Historical perspectives, mechanistic pathways and clinical translation for targeted cancer therapy. Signal Transduct Target Ther. 6 (1), 254(2021).
- O'Connor, M. J. Targeting the DNA damage response in cancer. Mol Cell. 60 (4), 547-560 (2015).
- Lord, C. J., Ashworth, A. The DNA damage response and cancer therapy. Nature. 481 (7381), 287-294 (2012).
- Carusillo, A., Mussolino, C. DNA Damage: From threat to treatment. Cells. 9 (7), 1665(2020).
- Li, X., Heyer, W. D. Homologous recombination in DNA repair and DNA damage tolerance. Cell Res. 18 (1), 99-113 (2008).
- Chang, H., Pannunzio, N. R., Adachi, N., Lieber, M. R. Non-homologous DNA end joining and alternative pathways to double-strand break repair. Nat Rev Mol Cell Biol. 18 (8), 495-506 (2017).
- Andratschke, N., et al. European society for radiotherapy and oncology and European organization for research and treatment of cancer consensus on re-irradiation: Definition, reporting, and clinical decision making. Lancet Oncol. 23 (10), e469-e478 (2022).
- Olivares-Urbano, M. A., Grinan-Lison, C., Marchal, J. A., Nunez, M. I. CSC radioresistance: A therapeutic challenge to improve radiotherapy effectiveness in cancer. Cells. 9 (7), 1651(2020).
- Schaue, D., McBride, W. H. Opportunities and challenges of radiotherapy for treating cancer. Nat Rev Clin Oncol. 12 (9), 527-540 (2015).
- Allen, C., Her, S., Jaffray, D. A. Radiotherapy for cancer: Present and future. Adv Drug Deliv Rev. 109, 1-2 (2017).
- Xue, C., Greene, E. C. DNA repair pathway choices in CRISPR-Cas9-Mediated genome editing. Trends Genet. 37 (7), 639-656 (2021).
- Tzelepis, K., et al. A CRISPR dropout screen identifies genetic vulnerabilities and therapeutic targets in acute myeloid leukemia. Cell Rep. 17 (4), 1193-1205 (2016).
- Rottner, A. K., et al. A genome-wide CRISPR screen identifies CALCOCO2 as a regulator of beta cell function influencing type 2 diabetes risk. Nat Genet. 55 (1), 54-65 (2023).
- Chen, M., et al. CRISPR-Cas9 for cancer therapy: Opportunities and challenges. Cancer Lett. 447, 48-55 (2019).
- Gupta, D., et al. CRISPR-Cas9 system: A new-fangled dawn in gene editing. Life Sci. 232, 116636(2019).
- Li, B., et al. Genome-wide CRISPR screen identifies host dependency factors for influenza A virus infection. Nat Commun. 11 (1), 164(2020).
- Hung, K. L., et al. Targeted profiling of human extrachromosomal DNA by CRISPR-CATCH. Nat Genet. 54 (11), 1746-1754 (2022).
- Bhattacharya, D., Van Meir, E. G. A simple genotyping method to detect small CRISPR-Cas9 induced indels by agarose gel electrophoresis. Sci Rep. 9 (1), 4437(2019).
- Razavi, Z., Soltani, M., Souri, M., Van Wijnen, A. J. CRISPR innovations in tissue engineering and gene editing. Life Sci. 358, 123120(2024).
- Wang, W., et al. A genome-wide CRISPR-based screen identifies KAT7 as a driver of cellular senescence. Sci Transl Med. 13 (575), eabd2655(2021).
- Xu, X., et al. Engineered miniature CRISPR-Cas system for mammalian genome regulation and editing. Mol Cell. 81 (20), 4333-4345 (2021).
- Huang, M., et al. FACS-based genome-wide CRISPR screens define key regulators of DNA damage signaling pathways. Mol Cell. 83 (15), 2810-2828 (2023).
- Feng, J., et al. Genome-wide CRISPR screen identifies synthetic lethality between DOCK1 inhibition and metformin in liver cancer. Protein Cell. 13 (11), 825-841 (2022).
- Saurat, N., et al. Genome-wide CRISPR screen identifies neddylation as a regulator of neuronal aging and AD neurodegeneration. Cell Stem Cell. 31 (8), 1162-1174 (2024).
- Du, Y., Liu, Y., Hu, J., Peng, X., Liu, Z. CRISPR/Cas9 systems: Delivery technologies and biomedical applications. Asian J Pharm Sci. 18 (6), 100854(2023).
- Shi, J., et al. A genome-wide CRISPR screen identifies WDFY3 as a regulator of macrophage efferocytosis. Nat Commun. 13 (1), 7929(2022).
- Liu, N., Olson, E. N. CRISPR modeling and correction of cardiovascular disease. Circ Res. 130, (2022).
- Feng, X., et al. Genome-wide CRISPR screens using isogenic cells reveal vulnerabilities conferred by loss of tumor suppressors. Sci Adv. 8 (19), 6638(2022).
- Ghaemi, A., et al. CRISPR-Cas9 genome editing delivery systems for targeted cancer therapy. Life Sci. 267, 118969(2021).
- Derry, W. B. CRISPR: Development of a technology and its applications. FEBS J. 288 (2), 358-359 (2021).
- Sharma, G., Sharma, A. R., Bhattacharya, M., Lee, S. S., Chakraborty, C. CRISPR-Cas9: A preclinical and clinical perspective for the treatment of human diseases. Mol Ther. 29 (2), 571-586 (2021).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。