Method Article
الدوائر الرقمية الجينية القائمة على أنظمة CRISPR-Cas والبروتينات المضادة ل CRISPR
* These authors contributed equally
In This Article
Summary
تم دمج أنظمة CRISPR-Cas والبروتينات المضادة ل CRISPR في مخطط البوابات المنطقية في Saccharomyces cerevisiae. أظهرت الدوائر المنطقية الصغيرة الجديدة أداء جيدا وعمقت فهم كل من عوامل النسخ المستندة إلى dCas9 / dCas12a وخصائص البروتينات المضادة لكريسبر.
Abstract
البوابات المنطقية للجينات الاصطناعية والدوائر الرقمية لها مجموعة واسعة من التطبيقات ، من التشخيص الطبي إلى الرعاية البيئية. يوفر اكتشاف أنظمة CRISPR-Cas ومثبطاتها الطبيعية - البروتينات المضادة ل CRISPR (Acrs) - أداة جديدة لتصميم وتنفيذ الدوائر الرقمية الجينية في الجسم الحي . هنا ، نصف بروتوكولا يتبع فكرة دورة الهندسة البيولوجية "Design-Build-Test-Learn" ويستخدم dCas9 / dCas12a جنبا إلى جنب مع Acrs المقابلة لها لإنشاء شبكات نسخ صغيرة ، بعضها يتصرف مثل البوابات المنطقية ، في Saccharomyces cerevisiae. تشير هذه النتائج إلى خصائص dCas9 / dCas12a كعوامل نسخ. على وجه الخصوص ، لتحقيق أقصى تنشيط للتعبير الجيني ، يحتاج dSpCas9 إلى التفاعل مع الحمض النووي الريبي سقالة مهندس يجمع نسخا متعددة من مجال تنشيط VP64 (AD). في المقابل ، يجب دمج dCas12a ، في المحطة C ، مع VP64-p65-Rta (VPR) AD القوي. علاوة على ذلك ، لا يتم تعزيز نشاط كل من بروتينات Cas عن طريق زيادة كمية sgRNA / crRNA في الخلية. تشرح هذه المقالة أيضا كيفية بناء بوابات منطقية بناء على تفاعل CRISPR-dCas-Acr. إن المجال المرتبط بالهرمونات المنصهر AcrIIA4 لمستقبلات هرمون الاستروجين البشري هو جوهر بوابة NOT التي تستجيب ل β-estradiol ، في حين أن AcrVAs التي يتم تصنيعها بواسطة مروج GAL1 المستحث يسمح بتقليد كل من بوابات YES و NOT مع الجالاكتوز كمدخل. في الدوائر الأخيرة ، أظهر AcrVA5 ، جنبا إلى جنب مع dLbCas12a ، أفضل سلوك منطقي.
Introduction
في عام 2011 ، اقترح الباحثون طريقة حسابية وطوروا برنامجا مطابقا للتصميم التلقائي لدوائر الجينات الاصطناعية الرقمية1. كان على المستخدم تحديد عدد المدخلات (ثلاثة أو أربعة) وملء جدول حقيقة الدائرة ؛ قدم هذا جميع المعلومات اللازمة لاشتقاق بنية الدائرة باستخدام تقنيات من الإلكترونيات. تمت ترجمة جدول الحقيقة إلى صيغتين منطقيتين عبر طريقة خريطة كارنو2. تتكون كل صيغة منطقية من عبارات تصف العمليات المنطقية (المجموع أو الضرب) بين (جزء من) مدخلات الدائرة ونفيها (الحرفية). الجمل ، بدورها ، إما تلخيصها (OR) أو ضربها (AND) لحساب خرج الدائرة. يمكن تحقيق كل دائرة وفقا لأي من الصيغتين المقابلتين لها: واحدة مكتوبة في شكل POS (حاصل ضرب المبالغ) والأخرى في تمثيل SOP (مجموع المنتجات). يتكون الأول من ضرب الجمل (أي البوابات المنطقية) التي تحتوي على مجموع منطقي للحرفيات. هذا الأخير ، على النقيض من ذلك ، هو مجموع الجمل حيث يتم ضرب الحرفيين.
يمكن تحقيق الدوائر الكهربائية ، على لوح التجارب ، عن طريق توصيل بوابات مختلفة معا فعليا. يسمح التيار الكهربائي بتبادل الإشارات بين البوابات ، مما يؤدي إلى حساب الإخراج.
في علم الأحياء ، الوضع أكثر تعقيدا. يمكن تحقيق البوابة المنطقية كوحدة نسخ (TU ؛ أي تسلسل "نهاية منطقة ترميز المروج" داخل الخلايا حقيقية النواة) ، حيث يتم تنظيم النسخ أو الترجمة (أو كليهما). وبالتالي ، يقوم نوعان على الأقل من الجزيئات بإنشاء أسلاك بيولوجية: بروتينات عامل النسخ والحمض النووي الريبي غير المشفروالمضاد للحساسية 1.
يتم تنظيم الدائرة الرقمية الجينية في طبقتين أو ثلاث طبقات من البوابات ، وهي: 1) طبقة الإدخال ، وهي مصنوعة من بوابات YES (عازلة) و NOT وتحول المواد الكيميائية المدخلة إلى جزيئات أسلاك. 2) الطبقة الداخلية ، والتي تتكون من العديد من TUs حيث توجد عبارات في الصيغة المنطقية المقابلة. إذا تم تصميم الدائرة وفقا لصيغة SOP ، فإن كل عبارة في الطبقة الداخلية ستنتج خرج الدائرة (على سبيل المثال ، مضان) في ما يسمى بمعمارية الإخراج الموزعة. إذا تم استخدام منتج صيغة المجموع (POS) ، فستكون هناك حاجة إلى طبقة نهائية 3) ، والتي ستحتوي على بوابة مضاعفة واحدة تجمع جزيئات الأسلاك من الطبقة الداخلية.
بشكل عام ، في البيولوجيا التركيبية ، يمكن تصميم العديد من المخططات المختلفة لنفس الدائرة. وهي تختلف في عدد ونوع كل من TUs وجزيئات الأسلاك. من أجل اختيار الحل الأسهل الذي سيتم تنفيذه في خلايا الخميرة ، يرتبط كل تصميم دائرة بدرجة تعقيد S ، تعرف بأنها

حيث يمثل A عدد المنشطات ، ويمثل R عدد المثبطات ، ويمثل a كمية جزيئات الحمض النووي الريبي المضادة للحساسية. إذا كانت المنشطات أو المثبطات غائبة عن الدائرة ، فإن مساهمتها في S تكون صفرا. لذلك ، من الصعب تحقيق مخطط دائرة في المختبر (ارتفاع S) عندما يتطلب عددا كبيرا من عوامل النسخ المتعامدة. وهذا يعني أنه يجب تصميم المنشطات والمثبطات الجديدة من جديد من أجل تحقيق الأسلاك الكاملة داخل الدوائر الرقمية. من حيث المبدأ ، يمكن تجميع البروتينات الجديدة المرتبطة بالحمض النووي باستخدام بروتينات إصبع الزنك3 ومستجيبات TAL4 كقوالب. ومع ذلك ، يبدو هذا الخيار شاقا للغاية ويستغرق وقتا طويلا. لذلك ، يجب على المرء أن يعتمد في الغالب على الحمض النووي الريبي الصغير وتنظيم الترجمة لوضع اللمسات الأخيرة على دوائر الجينات المعقدة.
في الأصل ، تم تطوير هذه الطريقة لتصنيع الدوائر الرقمية في البكتيريا. في الواقع ، في الخلايا حقيقية النواة ، بدلا من الحمض النووي الريبي المضاد للحساسية ، من الأنسب التحدث عن الحمض النووي الريبي الصغير (miRNAs) أو الحمض النووي الريبي الصغير المتداخل (siRNAs)5. ومع ذلك ، فإن مسار RNAi غير موجود في الخميرة S. cerevisiae. وبالتالي ، ينبغي للمرء أن يختار شبكات النسخ بالكامل. لنفترض أن الدائرة تحتاج إلى خمسة منشطات وخمسة مثبطات ؛ ستكون درجة تعقيدها S = 32. يمكن تقليل تعقيد الدائرة عن طريق استبدال عوامل النسخ ال 10 ب dCas96 واحد (Cas9 الذي يعاني من نقص النيوكلياز) مدمج في مجال تنشيط (AD). كما هو موضح في7 ، يعمل dCas9-AD كمثبط في الخميرة عند ربط المروج بين صندوق TATA و TSS (موقع بدء النسخ) وكمنشط عند الربط جيدا في المنبع لصندوق TATA. وبالتالي ، يمكن للمرء استبدال 10 عوامل نسخ ببروتين اندماج dCas9-AD واحد و 10 sgRNAs (RNAs دليل واحد) للحصول على درجة تعقيد إجمالية تبلغ S = 11. من السهل والسريع توليف عشرة sgRNAs ، بينما ، كما تم التعليق سابقا ، فإن تجميع 10 بروتينات سيتطلب عملا أطول وأكثر تعقيدا.
بدلا من ذلك ، يمكن للمرء استخدام اثنين من بروتينات dCas المتعامدة (على سبيل المثال ، dCas9 و dCas12a): أحدهما للاندماج في AD ، والآخر مجرد أو بالاشتراك مع مجال قمع. ستزداد درجة التعقيد بمقدار وحدة واحدة فقط (S = 12). وبالتالي ، فإن أنظمة CRISPR-dCas هي المفتاح لبناء دوائر رقمية جينية معقدة للغاية في S. cerevisiae.
تميز هذه الورقة بعمق كفاءة كل من المثبطات والمنشطات المستندة إلى dCas9 و dCas12a في الخميرة. تظهر النتائج أنها لا تتطلب كمية كبيرة من sgRNA لتحسين نشاطها ، لذلك يتم تجنب البلازميدات العرضية بشكل تفضيلي. علاوة على ذلك ، تكون المنشطات المستندة إلى dCas9 أكثر فاعلية عند استخدام سقالة RNA (scRNA) التي تقوم بتجنيد نسخ من VP64 AD. في المقابل ، يعمل dCas12a بشكل جيد عند دمجه في VPR AD القوي مباشرة. علاوة على ذلك ، يتطلب المروج المنشط الاصطناعي عددا متغيرا من المواقع المستهدفة ، اعتمادا على تكوين المنشط (على سبيل المثال ، ثلاثة عند استخدام dCas12a-VPR ، وستة ل dCas9-VP64 ، وواحد فقط مع dCas9 و scRNA). كقامع ، يبدو dCas12a أكثر وضوحا عند ربط منطقة الترميز بدلا من المروج.
ومع ذلك ، كعيب ، لا تتفاعل CRISPR-dCas9 / dCas12a مع المواد الكيميائية مباشرة. لذلك ، قد لا تكون ذات فائدة في طبقة الإدخال. لهذا السبب ، تم التحقيق في تصميمات بوابة منطقية بديلة تحتوي على بروتينات مضادة لكريسبر (Acrs). تعمل Acrs على (د) بروتينات Cas وتمنع عملها8. وبالتالي ، فهي وسيلة لتعديل نشاط أنظمة CRISPR-(d) Cas. تحلل هذه الورقة بدقة التفاعلات بين النوع الثاني Acrs و (d) Cas9 ، وكذلك النوع الخامس Acrs و (d) Cas12a في S. cerevisiae. نظرا لأن Acrs أصغر بكثير من بروتينات Cas ، فقد تم بناء بوابة NOT تستجيب للإستروجين β-استراديول عن طريق دمج مجال ربط الهرمونات لمستقبلات هرمون الاستروجين البشري 9-HBD (hER) -إلى AcrIIA4. إلى جانب ذلك ، تم تحقيق حفنة من بوابات نعم وليس التي عبرت عن dCas12a (-AD) بشكل أساسي و AcrVAs عند الحث مع الجالاكتوز. في الوقت الحاضر ، هذه البوابات تعمل فقط كدليل على المفهوم. ومع ذلك ، فإنها تمثل أيضا الخطوة الأولى نحو إعادة التفكير العميق في الخوارزمية لتنفيذ التصميم التلقائي الحسابي للدوائر الرقمية الجينية الاصطناعية في خلايا الخميرة.
Protocol
1. تصميم وبناء كاسيت التعبير sgRNA / crRNA
ملاحظة: هناك نوعان من أشرطة التعبير sgRNA / crRNA: SNR5210-أحادي المصطلح يتكون من مروج SNR52 المعتمد على RNA polymerase III ، وتسلسل sgRNA / crRNA ، وفاصل SUP4 ؛ آخر يختصر باسم RGR11 - يتكون من مروج ADH1 المعتمد على RNA polymerase II ، وهيكل RGR (RNA-ribozyme) الذي يحتوي على اثنين من الريبوزيمات (ريبوزيم-HH رأس المطرقة ، وفيروس التهاب الكبد دلتا ريبوزيم-HDV) وتسلسل sgRNA / crRNA بينهما ، وفاصل ADH1. تتكون متجانسات sgRNA التي توجه Cas9 من تسلسل فاصل والتكرار المباشرالمميز 12 ، في حين أن crRNA لبروتينات Cas12a تشتمل على التكرار المباشر متبوعا بتسلسل الفاصل13,14 (انظر الجدول التكميلي 1 لجميع تسلسلات الحمض النووي المستخدمة في هذه الدراسة).
- صمم تسلسل المباعد لتنشيط النسخ بوساطة Cas9 / Cas12a.
- استغل تسلسل مشغل lex البكتيري (المسمى lexOp) ليكون الموقع المستهدف 15,16 وأدخله في مروج CYC1 الذي يدفع التعبير عن بروتين الفلورسنت الأخضر المعزز بالخميرة (yEGFP)17. ومن ثم ، يتم تعريف تسلسل المباعد بواسطة lexOp المدرج ومكمل له.
- تحقق من تعامد تسلسل المباعد عبر أداة CRISPRDIRECT18.
- الصق تسلسل lexOp المحاط بتسلسل PAM في حقل النص ، وحدد تسلسل PAM على أنه NRG ل dCas9 و TTTV ل dCas12a ، وحدد الأنواع من القائمة المنسدلة على أنها الخميرة الناشئة (Saccharomyces cerevisiae) S288C Genome. انقر فوق تصميم. تأكد من عدم وجود موقع مستهدف مطابق في 20mer + PAM ولا في بحث 12mer + PAM.
- إنشاء كاسيت تعبير sgRNA / crRNA.
- استخدم تفاعل البوليميراز المتسلسل للهبوط لتضخيم تسلسل الحمض النووي للأجزاء البيولوجية القياسية ، مثل المروجين وتسلسلات الترميز والإنهايات.
- تحضير خليط تفاعل يحتوي على: 20-40 نانوغرام من قالب الحمض النووي ، 1 ميكرولتر من 10 ميكرومتر التمهيدي الأمامي (أي ، ot25 ، بناء البلازميد للتعبير sgRNA / crRNA) ، 1 ميكرولتر من التمهيدي العكسي 10 ميكرومتر (أي ، ot26 ، بناء بلازميد تعبير sgRNA / crRNA) ، 5 ميكرولتر من مزيج dNTP 2.5 mM ، 0.5 ميكرولتر من بوليميراز الحمض النووي ، 10 ميكرولتر من 5x عازلة تفاعل بوليميراز الحمض النووي ، والماء المقطر المزدوج (ddH2O) حتى حجم إجمالي قدره 50 ميكرولتر.
ملاحظة: راجع الجدول التكميلي 2 للحصول على قائمة بالبادئات المستخدمة في هذه الدراسة. - قم بتشغيل برنامج PCR للهبوط على جهاز تدوير حراري:
المرحلة 1: 98 درجة مئوية لمدة 30 ثانية.
المرحلة 2 مع 10 دورات: 98 درجة مئوية لمدة 10 ثوان ، 68 درجة مئوية لمدة 20 ثانية ، و 72 درجة مئوية لمدة 15 ثانية.
المرحلة 3 مع 25 دورة: 98 درجة مئوية لمدة 10 ثوان ، 59 درجة مئوية لمدة 20 ثانية ، و 72 درجة مئوية لمدة 15 ثانية.
المرحلة 4: 72 درجة مئوية لمدة 2 دقيقة.
أخيرا ، استمر عند 4 درجات مئوية حتى تبدأ تجارب المتابعة.
ملاحظة: تعتمد 68 درجة مئوية في المرحلة 2 و 59 درجة مئوية في المرحلة 3 على درجات حرارة انصهار كل من البادئات الأمامية والخلفية ، والتي تختلف من أزواج مختلفة من البادئات. يتم تحديد الوقت عند 72 درجة مئوية في المرحلتين 2 و 3 من خلال طول منتج تفاعل البوليميراز المتسلسل وسرعة بوليميراز الحمض النووي.
- تحضير خليط تفاعل يحتوي على: 20-40 نانوغرام من قالب الحمض النووي ، 1 ميكرولتر من 10 ميكرومتر التمهيدي الأمامي (أي ، ot25 ، بناء البلازميد للتعبير sgRNA / crRNA) ، 1 ميكرولتر من التمهيدي العكسي 10 ميكرومتر (أي ، ot26 ، بناء بلازميد تعبير sgRNA / crRNA) ، 5 ميكرولتر من مزيج dNTP 2.5 mM ، 0.5 ميكرولتر من بوليميراز الحمض النووي ، 10 ميكرولتر من 5x عازلة تفاعل بوليميراز الحمض النووي ، والماء المقطر المزدوج (ddH2O) حتى حجم إجمالي قدره 50 ميكرولتر.
- عزل منتجات تفاعل البوليميراز المتسلسل عن طريق هلام الكهربائي (100 فولت ، 30 دقيقة). تخلص من تسلسلات الحمض النووي من هلام الأغاروز عبر مجموعة استخراج هلام الحمض النووي (انظر جدول المواد).
ملاحظة: مطلوب هلام الأغاروز 0.8٪ للشظايا الأطول من 500 nt ، وهلام الأغاروز 1.5٪ للشظايا بين 100 nt و 500 nt ، وهلام الأغاروز 2٪ للشظايا الأقصر من 100 nt. - أدخل TU الذي يعبر عن sgRNA / crRNA في ناقل مكوك pRSII404 / 42419 ، والذي يحتوي على جين مقاومة الأمبيسلين وجين علامة التغذية الذاتية القابلة للاختيار من الخميرة TRP1.
- هضم ناقل المكوك عند 37 درجة مئوية لمدة 1 ساعة باستخدام إنزيمي التقييد SacI و Acc65I. تحضير خليط التفاعل عن طريق إضافة 5 ميكروغرام من ناقل المكوك ، والكميات الموصى بها من الإنزيمات ، ومخزن الهضم (وفقا لتعليمات الإنزيم) ، و ddH2O حتى حجم إجمالي قدره 30 ميكرولتر.
- تحقق من هضم ناقل المكوك عن طريق الرحلان الكهربائي الهلامي (انظر الخطوة الفرعية 1.3.2). ثم قم بتعطيل الإنزيمين عند 65 درجة مئوية لمدة 20 دقيقة.
- استخدم طريقة تجميع جيبسون 20 لإدخال منتجات تفاعل البوليميراز المتسلسل المنقى في ناقل المكوك المفتوح عن طريق السماح بدخول خليط الحمض النووي المتساوي المولي عند50 درجة مئوية لمدة 1 ساعة.
- قم بتحويل الخلايا المختصة بالإشريكية القولونية DH5α بخليط تفاعل جيبسون أعلاه عبر طريقة تحويل الصدمة الحرارية21. نقل خلايا الإشريكية القولونية المحولة إلى ألواح أجار لوريا بيرتاني (LB) التي تحتوي على الأمبيسلين (0.1 جم / لتر). ضع الألواح في الحاضنة عند 37 درجة مئوية واترك الخلايا تنمو بين عشية وضحاها.
- اختر أربع مستعمرات من صفيحة أجار LB واستزرعها بشكل منفصل طوال الليل عند 37 درجة مئوية في محلول LB يحتوي على الأمبيسلين (0.1 جم / لتر). بعد ذلك ، استخدم إجراء التحضير المصغر لاستخراج البلازميدات من خلايا الإشريكية القولونية 22.
- استخدم البادئات ot18 و ot19 (انظر الجدول التكميلي 2 لتسلسل oligo) لتسلسل وتأكيد وحدة النسخ المدرجة عبر طريقة سانجر23.
ملاحظة: في التجارب اللاحقة ، سيتم إدخال البلازميدات المبنية والمؤكدة في جينوم الخميرة عبر بروتوكول PEG / LiAc24.
- استخدم تفاعل البوليميراز المتسلسل للهبوط لتضخيم تسلسل الحمض النووي للأجزاء البيولوجية القياسية ، مثل المروجين وتسلسلات الترميز والإنهايات.
2. تصميم وبناء كاسيت تعبير RNA سقالة
ملاحظة: يتكون الحمض النووي الريبي دليل السقالة (scRNA) من تسلسل sgRNA وهياكل دبوس الشعر MS225. يتم استخدام نوعين من هياكل دبوس الشعر MS2 في هذا العمل: دبوس الشعر MS2 من النوع البري ، وبروتين غلاف MS2 من النوع F6 (MCP) aptamer-f6.
- توليف قالب scRNA قادر على استيعاب تسلسلات مباعدة مختلفة (على سبيل المثال ، pSNR52-spacer_DR(SpCas9)-2×MS2(wt+f6)-SUP4t).
ملاحظة: في هذه الدراسة ، تم تصنيع قالب scRNA بواسطة شركة تخليق الجينات (انظر جدول المواد). - صمم بادئات مناسبة (انظر الجدول التكميلي 2) لتشغيل تفاعل البوليميراز المتسلسل على تسلسلات المباعد اللازمة.
- اتبع الإجراءات الموجودة في الخطوة 1.3 لإنشاء شريط تعبير scRNA.
3. dSpCas9 الهندسة والتعبير بناء البلازميد
- الحصول على pTPGI_dSpCas9_VP64 البلازميد (انظر جدول المواد).
- بناء متجه المستقبل pRSII406-pGPD-ATG-XbaI-XhoI-CYC1t ، بناء على متجه المكوك pRSII406 ، عبر PCR الهبوط وطريقة تجميع جيبسون (راجع الخطوة 1.3). يوفر البلازميد مروجا تأسيسيا قويا - pGPD ، و terminator-CYC1t.
- هضم البلازميد pTPGI_dCas9_VP64 ومتجه المستقبل الذي تم إنشاؤه حديثا (5 ميكروغرام لمدة 1 ساعة أو 10 ميكروغرام للخطوة الليلية انظر 1.3.3.1 كمرجع) مع XbaI و XhoI عند 37 درجة مئوية. افصل ونقي جزء الإدراج ومتجه المستقبل كما في الخطوة 1.3.2.
- اربط جزء الإدخال المنقى وناقل المستقبل مع T4 DNA ligase عند 16 درجة مئوية لمدة 8 ساعات. تحضير محلول الربط عن طريق إضافة 50-100 نانوغرام من ناقل المستقبل المنقى ، وشظايا الهدف المنقى بكمية متساوية المولية مع ناقل المستقبل ، و 1.5 ميكرولتر من المخزن المؤقت T4 ، و 0.5 ميكرولتر من T4 ligase ، و ddH2O حتى إجمالي حجم 15 ميكرولتر.
- اتبع الخطوتين 1.3.3.3 و 1.3.3.4. بعد ذلك ، تأكد من صحة البلازميد الذي تم إنشاؤه حديثا عن طريق الهضم باستخدام XbaI و Xhol (37 درجة مئوية ، 1 ساعة) والرحلان الكهربائي الهلامي (انظر الخطوة 1.3.2).
4. dCas12a الهندسة وبناء البلازميد
- بناء البلازميدات التي تعبر عن dCas12a-AD.
- توليف اثنين من بروتينات dCas12a المحسنة لكودون الخميرة (denAsCas12a و dLbCas12a) محاطة بمواقع إنزيم تقييد BamHI و Xhol.
ملاحظة: في هذه الدراسة ، تم تصنيع بروتينات dCas12a المحسنة لكودون الخميرة بواسطة شركة تخليق الجينات (انظر جدول المواد). - قم ببناء متجه المستقبل pRSII406-promoter-ATG-NLS-GS-HIStag-GS-BamHI-sp-XhoI-AD-NLS-TAA-mTGUO1 عبر تفاعل البوليميراز المتسلسل للهبوط وطريقة تجميع جيبسون (انظر الخطوة 1.3) ، حيث يكون "المروج" إما pGPD أو pGAL1 ، ويمثل "sp" تسلسلا عشوائيا قصيرا ، و "AD" إما VPR أو VP64.
- أدخل كل بروتين dCas12a في نواقل الاستقبال التي تم إنشاؤها حديثا عن طريق الهضم مع BamHI و XhoI والربط مع T4 DNA ligase (راجع الخطوتين 3.3 و 3.4).
- توليف اثنين من بروتينات dCas12a المحسنة لكودون الخميرة (denAsCas12a و dLbCas12a) محاطة بمواقع إنزيم تقييد BamHI و Xhol.
- بناء البلازميدات التي تعبر عن dCas12a العارية.
- قم ببناء متجه متقبل لأشرطة التعبير المستحثة بالجالاكتوز ل dCas12a ك pRSII406-Acc651-pGAL1-ATG-NLS-GS-HIStag-GS-BamHI-sp-XhoI-GS-NLS-TAA-CYC1t باستخدام تفاعل البوليميراز المتسلسل الهبوطي وطريقة تجميع جيبسون (انظر الخطوة 1.3).
- هضم البلازميدات التي تحتوي على بروتينات dCas12a وناقل المستقبل أعلاه مع BamHI و Xhol ، ثم ربطها ب T4 DNA ligase للحصول على البلازميد pRSII406-pGAL1-Acc651-ATG-NLS-GS-HIStag-GS-BamHI-dCas12a-XhoI-GS-NLS-TAA-CYC1t (راجع الخطوتين 3.3 و 3.4).
- هضم البلازميد الذي تم الحصول عليه في الخطوة 4.2.2 مع Acc65I و BamHI ، ثم استبدل pGAL1 ب pGPD عبر PCR الهبوط وطريقة تجميع جيبسون لبناء pRSII406-pGPD-ATG-NLS-GS-HIStag-GS-BamHI-dCas12a-XhoI-GS-NLS-TAA-CYC1t.
5. هندسة البروتين المضادة لكريسبر وبناء البلازميد
ملاحظة: تم استخدام ثلاثة أنواع من المروجين لدفع تعبير Acrs: مروج محفز - pGAL1 ، وأربعة مروجين تأسيسيين للخميرة - pGPD ، و pACT1 ، و pTEF1 ، و pTEF2 ، ومروج تأسيسي اصطناعي -genCYC1t_pCYC1noTATA 26.
- احصل على البلازميدات التي تحتوي على تسلسلات النوع الثاني Acrs (AcrIIA2 و AcrIIA427 و AcrIIA5 28) والنوع V-A Acrs (AcrVA1 و AcrVA4 و AcrVA529) من شركة تخليق الجينات.
- بناء البلازميدات على أساس ناقل المكوك pRSII403 للتعبير عن Acrs.
- إنشاء أشرطة تعبير AcrIIA.
ملاحظة: استخدم تفاعل البوليميراز المتسلسل عند الهبوط لتضخيم أربعة مروجين مختلفين (أي pGPD و pACT1 و pTEF2 و genCYC1t_pCYC1noTATA) والأنواع الثلاثة من AcrIIA واثنين من المنهيات (ADH1t و CYC1t). قم ببناء سلسلة من TUs التي تعبر عن AcrIIAs ، تحت مروجين مختلفين ، عبر طريقة تجميع جيبسون (راجع الخطوة 1.3). - إنشاء أشرطة تعبير AcrVA.
- توليف تسلسل المستقبل: ATG-FLAGtag-GS-BamHI-sp-XhoI-NLS-GS-NLS-TAA-Tsynth6 ، حيث "sp" هو تسلسل عشوائي سيتم استبداله ب AcrVAs لاحقا.
ملاحظة: في هذه الدراسة ، تم تصنيع تسلسلات المستقبل بواسطة شركة تخليق الجينات (جدول المواد). - قم بتجميع متجهات المستقبل pRSII403-promoter-ATG-FLAGtag-GS-BamHI-sp-XhoI-NLS-GS-NLS-TAA-Tsynth6 ، حيث يكون "المروج": pGAL1 و pTEF1 و genCYC1t_pCYC1noTATA. استخدم طريقة تجميع جيبسون (راجع الخطوة 1.3).
- أدخل كل من AcrVA الثلاثة في متجه المستقبل الموصوف في الخطوة 5.2.2.2 (عبر تفاعل البوليميراز المتسلسل عند الهبوط وطريقة تجميع جيبسون [انظر الخطوة 1.3]) لبناء مجموعة من البلازميدات المنتجة ل AcrVAs.
- توليف تسلسل المستقبل: ATG-FLAGtag-GS-BamHI-sp-XhoI-NLS-GS-NLS-TAA-Tsynth6 ، حيث "sp" هو تسلسل عشوائي سيتم استبداله ب AcrVAs لاحقا.
- إنشاء أشرطة تعبير AcrIIA.
- مزيد من الهندسة AcrIIA4 من خلال توسيع تسلسلها مع تسلسل رابط GS و HBD (hER). هذا يسمح ببناء دائرة مستجيبة β استراديول.
- استخدم تفاعل البوليميراز المتسلسل عند الهبوط للحصول على أجزاء GS-HBD و AcrIIA4 بشكل منفصل (انظر الخطوة 1.3.1).
- ضع AcrIIA4 و GS-HBD ومتجه المكوك في خليط جيبسون وقم ببناء البلازميد الكامل عبر طريقة جيبسون (انظر الخطوة 1.3.3).
6. كشف crRNA: RT-qPCR وتصميم الاشعال
ملاحظة: تم الكشف عن crRNA عبر RT-qPCR ، والذي تم تنظيمه في ثلاث خطوات.
- قم بإجراء استخراج الحمض النووي الريبي وتنقيته من خلايا الخميرة عبر مجموعة الحمض النووي الريبي.
- زراعة خلايا الخميرة بين عشية وضحاها في 2 مل من الوسط الكامل المحدد الاصطناعي (SDC ، حجم 1 لتر: 20 جم من الجلوكوز ، 2 جم من مزيج AA ، 6.7 جم من YNB ، 396 مجم من الليوسين ، 79.2 مجم من التربتوفان ، 79.2 مجم من الهستيدين ، و 79.2 مجم من اليوراسيل) باستخدام صفيحة 24 بئرا (240 دورة في الدقيقة ، 30 درجة مئوية).
- في الصباح ، قم بتخفيف زراعة الخلايا (1: 100) إلى 2 مل من SDC الطازج واستمر في زراعة خلايا الخميرة عند 30 درجة مئوية ، 240 دورة في الدقيقة ، لمدة 4 ساعات أخرى.
- احصد 2 مل بالكامل من محلول الخلية وأجهزة الطرد المركزي عند 20238 × جم لمدة دقيقتين. قم بإزالة المادة الطافية بعناية لأن حبيبات الخلية صغيرة.
- استخراج الحمض النووي الريبي من خلايا الخميرة باستخدام مجموعة الحمض النووي الريبي.
- تحقق من جودة الحمض النووي الريبي.
- إعداد هلام الأغاروز 1 ٪. امزج 5 ميكرولتر من كل عينة من الحمض النووي الريبي مع 1 ميكرولتر من صبغة تحميل الحمض النووي. ثم قم بتحميل الخليط على الجل وتشغيله.
- تأكد من وجود شريطين واضحين عند ~ 4000 nt و ~ 2000 nt ، يقابلان الحمض النووي الريبوزي الريبوسومي (25S / 18S) ، على الجل. يمكن رؤية نطاق غير واضح آخر عند ~ 80 nt ل tRNA.
- استخدم عينات الحمض النووي الريبي على الفور لتخليق cDNA أو قم بتخزينها في -80 درجة مئوية لاستخدامها في المستقبل.
- النسخ العكسي: استخدم طريقة الحلقة الجذعية 30 لتشكيل الشريط الأول من cDNA المقابل ل crRNA (40 nt). بالنسبة للنسخ العكسي ل sgRNA (حوالي 100 nt) ، فإن الإجراء هو نفسه الخاص بتوليف cDNA للجين المرجعي ACT1.
ملاحظة: تختلف طريقة النسخ العكسي ل crRNA عن تلك المستخدمة مع sgRNA و ACT1 mRNA. نظرا لأن crRNA قصير جدا ، فقد تم التعامل معه على أنه microRNA واستخدم طريقة النسخ العكسي microRNA (نهج حلقة الجذع) للحصول على cDNA المقابل. تم استخدام مجموعتين من مجموعات تخليق cDNA (مجموعة حلقة جذعية ل crRNA ومجموعة نسخ عكسي معتادة لجين ACT1) في القياس الكمي crRNA . تم استخدام نفس الكمية من الحمض النووي الريبي في المجموعتين (انظر جدول المواد) لجعل نتائج التجربة قابلة للمقارنة. تم تصميم التمهيدي المستخدم مع مجموعة الحلقة الجذعية وفقا لتسلسل الحلقة الجذعية والنيوكليوتيدات الستة الأخيرة في نهاية 3 'من crRNA (للاطلاع على تسلسل الحلقة الجذعية والتمهيدي ، انظر الجدول التكميلي 2).- طريقة حلقة الجذعية للنسخ العكسي crRNA
- أخرج قالب الحمض النووي الريبي والتمهيدي والمخازن المؤقتة من الفريزر واتركها تذوب على الجليد.
- إزالة الحمض النووي الجينومي: أولا ، قم بإعداد 10 ميكرولتر من خليط التفاعل وفقا لتعليمات المجموعة. ضعي الخليط في جهاز تدوير حراري على حرارة 42 درجة مئوية لمدة 2 دقيقة. أخيرا ، انقله إلى الجليد.
- تخليق شريط cDNA الأول: تحضير 20 ميكرولتر من خليط التفاعل بإضافة 10 ميكرولتر من الخليط من الخطوة 6.2.1.2 ، 1 ميكرولتر من التمهيدي للحلقة الجذعية (تركيز 2 ميكرومتر) ، 2 ميكرولتر من محلول تفاعل RT 10x ، 2 ميكرولتر من مزيج إنزيم النسخ العكسي (يحتوي على النسخ العكسي) ، و 5 ميكرولتر من H2O الخالي من RNase.
- ضع خليط التفاعل في جهاز تدوير حراري وقم بتشغيل البرنامج التالي: 25 درجة مئوية لمدة 5 دقائق ، و 50 درجة مئوية لمدة 15 دقيقة ، و 85 درجة مئوية لمدة 5 دقائق. استخدم المنتج لتفاعل qPCR على الفور أو قم بتخزينه في -80 درجة مئوية.
- sgRNA و ACT1 mRNA النسخ العكسي
- التفاعل الأول: تحضير خليط 13 ميكرولتر يتكون من مزيج التمهيدي ، ومزيج dNTP ، وقالب الحمض النووي الريبي (50 بيكوغرام -5 ميكروغرام) ، والماء الخالي من RNase (بصرف النظر عن قالب الحمض النووي الريبي ، وكلها مقدمة من المجموعة) ، وفقا لتعليمات المجموعة. استخدم 1 ميكروغرام من قالب الحمض النووي الريبي. ضعي الخليط في جهاز تدوير حراري على حرارة 70 درجة مئوية لمدة 10 دقائق.
- التفاعل الثاني (تخليق cDNA): قم بإعداد خليط التفاعل عن طريق إضافة الكواشف (كما هو موضح في تعليمات المجموعة) إلى 13 ميكرولتر من محلول التفاعل الأول حتى الحجم النهائي 20 ميكرولتر. ضع الخليط في جهاز تدوير حراري لمدة 15 دقيقة عند 50 درجة مئوية ثم لمدة 5 دقائق إضافية عند 85 درجة مئوية. استخدم المنتج لتفاعل qPCR على الفور أو قم بتخزينه في -80 درجة مئوية.
- طريقة حلقة الجذعية للنسخ العكسي crRNA
- مجموعة SYBR ل qPCR: كشف قيمة CT
ملاحظة: تم إصلاح التمهيدي العكسي المستخدم في qPCR ل crRNA لأنه يتوافق مع المكمل العكسي لتسلسل الحلقة الجذعية (انظر الجدول التكميلي 2). التمهيدي الأمامي ، في المقابل ، متغير ويعتمد على تسلسل crRNA. تم تصميم البادئات الأمامية والخلفية ل sgRNA و ACT1 mRNA qPCR عند https://www.ncbi.nlm.nih.gov/tools/primer-blast/. يتم اختيار اثنين من البادئات عندما لا يزيد الفرق بين درجات حرارة انصهارها عن 2 درجة مئوية (انظر الجدول التكميلي 2). يتم قياس كل عينة في ثلاث نسخ متماثلة.- قم بإعداد خليط تفاعل qPCR وفقا لتعليمات الشركة المصنعة لمجموعة SYBR.
- اضبط برنامج qPCR التالي في جهاز PCR في الوقت الفعلي.
مرحلة ما قبل الحضانة: 10 دقائق عند 95 درجة مئوية.
مرحلة تفاعل البوليميراز المتسلسل: 15 ثانية عند 95 درجة مئوية ، تليها 34 ثانية عند 55 درجة مئوية. اضبط دورة مرحلة PCR على 45 مرة. مرحلة الانصهار: 10 ثوان عند 95 درجة مئوية ، تليها 60 ثانية عند 65 درجة مئوية ، وأخيرا 1 ثانية عند 97 درجة مئوية. - احسب قيم تعبير mRNA النسبية عبر صيغة Pfaffl31.
7. التألق المناعي للكشف عن بروتينات كاس
ملاحظة: يتم دمج بروتينات Cas (CasP) في His_tag.
- تحضير خلايا الخميرة
- اختر بعض خلايا الخميرة باستخدام حلقة معقمة واستزرعها في 5 مل من الوسط الغني ب YPD طوال الليل عند 240 دورة في الدقيقة عند 30 درجة مئوية. في الصباح ، أضف 500 ميكرولتر من محلول الخلية إلى 20 مل من YPD الطازج وقم بزراعتها عند 240 دورة في الدقيقة عند 30 درجة مئوية حتى يصل OD600 إلى 0.6.
- خذ 5 مل من الثقافة واخلطها مع 500 ميكرولتر من 37٪ فورمالديهايد. دع الخليط يبقى في درجة حرارة الغرفة (RT) لمدة 10 دقائق. حصاد الخلايا عن طريق الطرد المركزي عند 1500 × غرام لمدة 5 دقائق.
- قم بإزالة المادة الطافية وأعد تعليق الخلايا ب 1 مل من المخزن المؤقت للتثبيت (0.1 M KH 2 PO4 ، 0.5 M MgCl2، 3.7٪ فورمالديهايد ، درجة الحموضة = 6.5). احتفظ بمحلول الخلية في RT لمدة 20 دقيقة.
- جهاز طرد مركزي محلول الخلية عند 1500 × جم لمدة 5 دقائق. تخلص من المادة الطافية وأعد تعليق الخلايا في 1 مل من محلول الغسيل (0.1 M KH 2 PO 4 ،1.2M سوربيتول ، درجة الحموضة = 6.5) مكملة ب 4 ميكرولتر من بيتا ميركابتوإيثانول و4 ميكرولتر من محلول المحللة (5 مجم / مل). ضع محلول الخلية في حاضنة على حرارة 37 درجة مئوية لمدة 20 دقيقة.
- قم بطرد محلول الخلية عند 1500 × جم لمدة 5 دقائق وتخلص من المادة الطافية. اغسل حبيبات الخلية مرتين باستخدام 1 مل من PBS عن طريق الطرد المركزي (1,500 × جم لمدة 5 دقائق).
- أعد تعليق الخلايا في 100 ميكرولتر من PBS بالإضافة إلى 0.05٪ Tween 20 وأضف 4 ميكرولتر من محلول BSA (10 مجم / مل). احتفظ بمحلول الخلية في RT لمدة 20 دقيقة.
- الحضانة مع الأجسام المضادة الأولية
- أضف الجسم المضاد الأساسي لعلامة Anti-His إلى الخليط في الخطوة 7.1.6 عند تخفيف 1: 400. الحفاظ على الحل في RT لمدة 2 ساعة.
- قم بطرد الخليط في الخطوة 7.2.1 عند 1500 × جم لمدة 5 دقائق وإزالة المادة الطافية. أضف 1 مل من PBST وأجهزة الطرد المركزي (1500 × جم) لمدة 5 دقائق لغسل حبيبات الخلية. كرر هذه العملية مرتين. أخيرا ، تخلص من المادة الطافية وقم بتعليق الخلايا في 100 ميكرولتر من 1x PBST.
- الكشف المجهري عن الخلايا
- قم بتركيب الخلايا على شريحة ؛ خذ 2 ميكرولتر من محلول الخلية من الخطوة 7.2.2 وضعه على شريحة زجاجية. قم بتغطيتها بغطاء غطاء.
- مراقبة الخلايا تحت المجهر مضان. قم بتشغيل مصدر ضوء الفلورسنت والمجهر والكمبيوتر. اكتب رقم مصدر ضوء الفلورسنت وافتح برنامج المجهر على الكمبيوتر.
- ضع الشريحة على مرحلة المجهر. اختر العدسة الموضوعية 40x وراقب الخلايا تحت الضوء الأخضر (550 نانومتر). حرك مقبض التركيز الخشن حتى يظهر محيط خلايا الخميرة. حرك مقبض التركيز البؤري الدقيق لتركيز الخلايا.
- كشف الخلايا باستخدام برنامج المجهر. أغلق مجال رؤية المجهر وانتقل إلى شاشة الكمبيوتر. انقر فوق Live ، وانتظر لمدة 3-5 ثوان ، وانقر فوق Capture لالتقاط صورة. احفظ الصورة.
- قم بإيقاف تشغيل الكمبيوتر والمجهر ومصدر ضوء الفلورسنت.
8. الحصول على البيانات: FACS
ملاحظة: يتم الكشف عن التألق الأخضر عن طريق قياس التدفق الخلوي (أي قياسات فرز الخلايا المنشطة بالتألق [FACS]). يتم استزراع خلايا الخميرة ، بشكل عام ، عند 30 درجة مئوية و 240 دورة في الدقيقة لإجراء تجارب FACS. ومع ذلك ، قد تتطلب الخلايا بعض الاحتياطات اعتمادا على محتواها الجيني. يجب زراعة الخلايا التي تحتوي على جين dCas12a-VPR (الذي يتحكم فيه المروج التأسيسي GPD ) لمدة 24 ساعة في محلول SDC. بعد ذلك ، يتم تخفيف الخلايا بنسبة 1: 100 في SDC الطازج وتنمو لمدة 12 ساعة أخرى قبل قياس شدة التألق. تتطلب الخلايا المعدلة باستخدام جين AcrIIA4-HBD (hER) التخفيف أيضا. علاوة على ذلك ، يجب التحكم في OD600 . أولا ، يسمح للخلايا بالنمو في SDC بين عشية وضحاها (أكثر من 14 ساعة). في الصباح ، يتم قياس OD600 . ثم يتم تخفيف الثقافة في SDC ، مع تزويدها بتركيز متنوع من β-استراديول ، وصولا إلى OD600 = 0.1. قبل تجارب FACS ، تزرع الخلايا لمدة 7 ساعات أخرى بحيث يصل OD600 إلى 0.8-1.0. تزرع الخلايا التي تعبر عن dCas9-VP64 أو dCas12a-VP64 في SDC لمدة 20-24 ساعة دون تخفيف ومزيد من النمو قبل القياسات في جهاز FACS.
- قم بتشغيل جهاز FACS قبل 20 دقيقة من القياسات لتسخين الليزر.
- تحضير العينات (التخفيف): خلط 20 ميكرولتر من ثقافة الخلية مع 300 ميكرولتر من ddH2O.
- قم بتشغيل برنامج FACS على الكمبيوتر المتصل بجهاز FACS وقم بإنشاء تجربة جديدة. اضبط معلمات القياس (على سبيل المثال ، FSC (SSC) -A / H / W والرسم البياني).
- حدد المرشح وفقا للأطوال الموجية للإثارة والانبعاث للعينات. على سبيل المثال ، البروتين المستهدف هنا هو yEGFP ، الذي تبلغ أطواله الموجية للإثارة والانبعاث 488 نانومتر و 507 نانومتر على التوالي. لذلك ، حدد مرشح FITC أو GFP (الطول الموجي للإثارة: 488 نانومتر ؛ الطول الموجي للانبعاث: 527/32 نانومتر). قم بتعيين رقم خلية الاستحواذ إلى 10000.
- اضبط جهد مرشح FITC عن طريق قياس شدة حبات الفلورسنت. التأكد من أن الفرق النسبي في شدة الخرز بين تجربتين متتاليتين لا يتجاوز 5٪.
- اغسل الغسالة ب ddH2O لبضع ثوان لإزالة أي خرز زائد محتمل.
- قياس شدة مضان العينة. انقر فوق معاينة وانتظر 3-5 ثوان لاستقرار حقن العينة. أخيرا ، انقر فوق اكتساب.
- قم بقياس الخرزات مرة أخرى في نهاية التجربة. الجهد هو الذي تم استخدامه أثناء قياس الخرز الأولي (انظر الخطوة 8.4 ، 438-441 فولت). تحقق مما إذا كان الفرق النسبي بين قياسات الخرزتين يتجاوز 5٪.
- تصدير بيانات FACS كملفات FCS.
- تحليل الملفات FCS مع برنامج R ستوديو.
9. تحليل البيانات
ملاحظة: استخدم حزمة الموصلات الحيوية Flowcore R32 داخل استوديو R. تم تحليل ملفات FCS باستخدام برنامج نصي مكتوب بلغة R.
- افتح استوديو R وقم بتحميل تحليل البرنامج النصي Bdverse. R لتحليل ملفات FCS.
- تعيين اسم التجربة (ename) والدليل (المسار) حيث توجد ملفات FCS
مخزنة (dir_d) ، وحيث سيتم إنشاء ملفات النتائج (dir_r). - اضبط القناة الفلورية. على سبيل المثال ، select_ch = "GPA-A" إذا تم قياس التألق الأخضر.
- تعيين عدد العينات التي تم قياسها (s_num).
- اضبط أبعاد كل مخطط نقطي (sxlim ، sylim). اضبط الحد الأقصى لطول المحور x و y للمخططات الشريطية ومخططات المربع (xlimit ، ylimit). يجب أن يكون xlimit أكبر من أو يساوي s_num.
- اختر طريقة البوابات عن طريق إزالة # من الخطوط المقابلة.
ملاحظة: morphGate هي طريقة بوابة تلقائية يتم تنفيذها بواسطة البرنامج النصي (على سبيل المثال ، يتم التعرف على منطقة المخططات النقطية حيث تكون الخلايا أكثر كثافة وتحديدها بواسطة البرنامج). يتطلب polygonGate و rectangleGate إلقاء نظرة على المخططات النقطية وتحديد إما رؤوس المضلع أو جانب المستطيل الذي يحتضن المنطقة التي تقع فيها معظم الخلايا. - حدد كائن flowSet ببوابة ، المقابلة لطريقة البوابة المختارة. حدد دقة الرسم النقطي (الدقة ؛ يجب أن تساوي 256 على الأقل).
- قم بإلغاء تعليق flt_low <- filter_low (sp) لإزالة القياسات التي يكون فيها التألق سالبا. قم بإلغاء التعليق flt_sp <- filter (flt_lw) لإزالة القيم المتطرفة بسبب تجارب أخرى.
- اضغط على المصدر وقم بتشغيل البرنامج النصي. يتم إنشاء جميع الملفات التي تحتوي على نتائج التحليل في dir_r.
النتائج
تعبير sgRNA / crRNA بواسطة مروج من نوع بوليميراز الحمض النووي الريبي من النوع الثالث
أولا ، تناول هذا العمل هندسة دائرة تنشيط النسخ (الدائرة 1) الموضحة في الشكل 1 أ. احتوى على ثلاثة مكونات أساسية: 1) الترميز الجيني ل yEGFP (المراسل) ، والذي سبقته سلسلة من المروجين الاصطناعيين المختلفين الذين قدموا مواقع مستهدفة ل dCas9 / dCas12a-AD. 2) نسخة محسنة لكودون الخميرة من dCas9 أو dCas12a مدمجة في مجال تنشيط (VP64 و VPR ، على التوالي) وتحتوي إما على تسلسل واحد أو اثنين من تسلسلات التوطين النووي (NLSs). تم إنتاج كل من بروتينات dCas بواسطة مروج تأسيسي قوي - pGPD. و 3) تسلسل sgRNA / crRNA الذي وجه dCas9 / dCas12a-AD إلى المواقع المستهدفة. تم تصور كفاءة التنشيط للمنشطات القائمة على dCas9 / dCas12a وانعكست من خلال شدة التألق للمراسل (تم قياسها من خلال تجارب FACS).
تم اختبار بروتين dCas9 واحد (dSpCas9) وبروتينين dCas12a (denAsCas12a و dLbCas12a). تم تحقيق تنشيط 3.36 أضعاف مع تمديد dSpCas9 ، في نهايته C ، مع VP64 AD وربط مروج اصطناعي من المنبع من yEGFP يحتوي على ست نسخ من موقع lexOp المستهدف. تم وضع sgRNA في ناقل مكوك تكاملي ونسخه بواسطة مروج SNR52 المعتمد على بوليميراز الحمض النووي الريبي III (تكوين SNR52i ، انظر الشكل 1B). في حالة dCas12a ، أعاد denAsCas12a-VPR أعلى تنشيط (4.45 أضعاف) من مروج اصطناعي مع ثلاثة عوامل عندما تم التعبير عن crRNA عبر تكوين SNR52i (الشكل 1C). في ظل نفس الظروف ، حقق dLbCas12a-VPR أفضل تحسين مضان (3.21 أضعاف) (الشكل 1D). وتجدر الإشارة إلى أن مصطلح المقارنة، في كل تجربة، هو دائرة لا يمكن ل sgRNA/crRNA أن تربط مشغلي القانون.
البلازميدات متعددة النسخ ليست ضرورية
تم استبدال شريط تعبير SNR52i sgRNA بهيكل RGR يعبر عنه مروج قوي إلى حد ما pADH1. ومع ذلك ، في كل من حالات dCas9 و dCas12a ، بدا التنشيط في وجود sgRNA / crRNA الناتج عن الانقسام الذاتي ل RGR مشابها أو حتى أقل من ذلك الذي تم تحقيقه باستخدام sgRNA / crRNA المنتج عبر SNR52i ، على الرغم من حقيقة أن pSNR52 كان يعتبر محفزا ضعيفا (انظر الشكل 1C ، D للحصول على النتائج الأولى التي تم الحصول عليها باستخدام dCas12a).
لمزيد من استكشاف العلاقة بين كمية sgRNA / crRNA وكفاءة التنشيط ، تم إدخال نظامي التعبير sgRNA / crRNA في بلازميد عرضي ، والذي يمكن أن تمتصه الخلية في 10-40 نسخة وتوليد كمية أكبر من sgRNA / crRNA. كما هو موضح في الشكل 2 أ ، كان التنشيط بواسطة crRNA الموجود على بلازميد تكاملي (إما SNR52i أو RGRi) أعلى بمقدار 1.4 إلى 2.4 مرة من ذلك الذي تم تحقيقه عندما تم التعبير عن نفس crRNA بواسطة بلازميد عرضي (إما SNR52m أو RGRm). تم تأكيد هذا الاتجاه من قبل sgRNA. في هذه الحالة ، يضمن البلازميد التكاملي تنشيطا أعلى بمقدار 1.1 إلى 1.5 مرة (الشكل 2 ب). لاستبعاد أن النتائج كانت ناجمة عن فقدان البلازميدات العرضية ، تم إجراء RT-qPCR لتحديد الوفرة النسبية sgRNA / crRNA في الجسم الحي. تحققت النتائج ، في الشكل 2C ، D ، من أن الناقل العرضي أنتج مستوى أعلى بكثير من sgRNA / crRNA من المتجه التكاملي ، بغض النظر عن نظام التعبير (RGR أو SNR52). أظهرت هذه النتائج أن نظام SNR52 يمكن أن يعمل بشكل أفضل من نظام RGR ، وأن كمية أكبر من sgRNA / crRNA في الخلية لم تضمن تنشيطا أعلى بواسطة نظام CRISPR-Cas. لذلك ، لا ينبغي استخدام البلازميدات العرضية في بناء الدوائر الرقمية الجينية حيث يتم استخدام dCas9 / dCas12a-AD.
سقالة هندسة الحمض النووي الريبي
تم تصميم scRNA عن طريق توسيع تسلسل sgRNA مع هياكل دبوس الشعر MS2 التي سمحت بتجنيد VP64 AD عند دمجها في بروتين غلاف MS2 (MCP ، انظر الشكل 3A). بهذه الطريقة ، لم تكن هناك حاجة إلى هندسة أو تعديلات على dCas9. تمت تجربة نوعين مختلفين من MS2: WT و F6. أعطى scRNA الذي يحتوي على دبوس شعر MS2 واحد -1×MS2 (بالوزن) و 1×MS2 (f6) - تنشيطا بمقدار 5.27 ضعفا و 4.34 ضعفا على التوالي. ومع ذلك ، فإن scRNA مع مزيج من دبابيس الشعر - 2×MS2 (wt + f6) - أعاد أعلى تنشيط عام في هذه الدراسة (7.54 أضعاف ، انظر الشكل 3B). أظهرت هذه النتائج أن هندسة scRNA كانت أكثر فعالية بكثير من دمج أي مجالات تنشيط إلى dSpCas9 مباشرة.
بوابة منطقية تعتمد على بروتينات Acr
لمزيد من التحكم في التنشيط النسخي وضبطه بواسطة أنظمة CRISPR-Cas ، تم تعديل الدائرة 1 بإدخال وحدة TU رابعة للتعبير عن البروتينات المضادة ل CRISPR (انظر الشكل 4A ل AcrIIAs والشكل 5A ل AcrVAs). بعد إظهار أن Acrs كانت فعالة ، في S. cerevisiae ، في مقارنة التنشيط بسبب dCas9 / dCas12a-AD ، تم بناء دائرة جديدة (انظر الشكل 5D) لاختبار عمل AcrVA على المثبطات المستندة إلى dCas12a (أظهر عمل سابق33 بالفعل أن AcrIIAs يمكن أن تمنع تنظيم الجينات المستندة إلى dCas9). عملت الشبكات الصغيرة الجديدة المحتوية على Acr كبوابات منطقية بسيطة (نعم و NOT) ، مما قد يؤدي إلى إعادة تصفيف طبقة الإدخال للدوائر الرقمية الجينية الاصطناعية الأكثر تعقيدا.
AcrIIA4 هو مثبط قوي ل dSpCas9
تم استخدام أربعة مروجين مختلفين يتمتعون بنقاط قوة متنوعة لدفع التعبير عن ثلاثة أنواع من AcrIIs-AcrIIA2 و AcrIIA4 و AcrIIA5. أظهرت النتائج أن AcrIIs الثلاثة عملت بطريقة تعتمد على الجرعة في S. cerevisiae. عندما تم التعبير عنها من قبل مروج قوي - pGPD - قاموا بتخفيض مستوى التألق الذي تم الوصول إليه بواسطة dSpCas9: scRNA_2× MS2 (wt + f6) -MCP-VP64 إلى 0.21 و 0.11 و 0.13 من قيمته ، على التوالي (الشكل 4 ب). نظرا لأن AcrIIA4 كان الوحيد الذي تسبب في تثبيط عالي للتعبير الفلوري حتى عندما يتم إنتاجه بواسطة genCYC1t_pCYC1noTATA محفز اصطناعي ضعيف - يمكننا أن نستنتج أن AcrIIA4 كان أقوى مثبط بين AcrIIs الثلاثة. بعد ذلك ، تم دمج HBD (ER) في المحطة C ل AcrIIA4 لبناء جهاز استشعار β-استراديول (انظر الشكل 4C). في وجود الإستروجين β-استراديول ، يمكن أن ينتقل AcrIIA4-HBD (ER) إلى النواة ثم يحيد وظيفة المنشط القائم على dSpCas9. يوضح منحنى المعايرة بالتحليل الحجمي في الشكل 4D أن الدائرة تتصرف مثل بوابة NOT بنسبة تشغيل / إيقاف تقترب من 2.3.
AcrVAs هي مثبطات لبروتينات dCas12a
تم تصميم وبناء بوابة NOT عن طريق إدخال كاسيت تعبير AcrVA الذي يقود pGAL1 في الدائرة 1. بهذه الطريقة ، يمكن أن يحدث تخليق AcrVA ، وما يترتب على ذلك من قمع dCas12a-AD ، عن طريق الجالاكتوز (الشكل 5A). كما هو موضح في الشكل 5B ، C ، أعاق AcrVA1 كلا من denAsCas12a و dLbCas12a كمنشطات عن طريق تقليل التعبير الفلوري من 19٪ إلى 71٪ ، اعتمادا على مخطط الدائرة. لا يمكن ل AcrVA4 و AcrVA5 ممارسة أي إجراء على denAsCas12a34. ومع ذلك ، فقد قاموا بتثبيط قوي للمنشطات المستندة إلى dLbCas12a عن طريق تقليل التعبير الفلوري بنسبة تصل إلى 84٪ (AcrVA5) و 82٪ (AcrVA4). بشكل عام ، تبين أن AcrVA5 هو الأكثر موثوقية ، من بين هذه AcrVAs الثلاثة ، في تثبيط المنشطات المستندة إلى dLbCas12a لأنه يضمن ، في دوائر مختلفة ، قمعا أعلى من 70٪.
عمل AcrVA1 يعتمد على التركيز
كما تمت دراسة العلاقة بين تركيز AcrVA في الجسم الحي وتأثيراتها المثبطة على كل من المنشطات والمثبطات القائمة على denAs / dLbCas12a. لهذا الهدف ، تم التعبير عن كل AcrVA تحت مروجين مختلفين: pGAL1 القوي ، pTEF1 متوسط القوة ، و genCYC1t_pCYC1noTATA الضعيف. كما هو موضح في الشكل 5E ، أظهر AcrVA1 تقلبات كبيرة في أدائه اعتمادا على المروج الذي قاد توليفه. يعمل AcrVA1 بشكل جيد فقط عند إنتاجه بواسطة pGAL1. تحت pTEF1 و genCYC1t_pCYC1noTATA ، أظهر AcrVA1 بعض القمع على dLbCas12a المجرد فقط. في المقابل ، بدا أن AcrVA4 و AcrVA5 أقل حساسية لتركيزهما ، خاصة عند التفاعل مع dLbCas12a العاري.
أظهرت هذه البيانات أن أداء AcrVA4 و AcrVA5 بشكل عام أفضل من AcrVA1 في تثبيط عوامل النسخ المستندة إلى dLbCas12a في S. cerevisiae. تجدر الإشارة إلى أن AcrVA5 لديه آلية عمل غريبة ، لأنه يعمل كإنزيم يعدل LbCas12a بشكل دائم. ومع ذلك ، كما ذكر أعلاه ، لا يمكن ل AcrVA5 (مع AcrVA4) التفاعل مع denAsCas12a.
عندما تم التعبير عن AcrVAs تحت pGAL1 ، تصبح الدوائر إما بوابات YES (تم دمج dCas12a في AD) أو بوابات NOT (bare dCas12a). بدا الأول عالي الأداء ، بينما بدا أن الأخير يعمل بشكل أفضل في وجود AcrVA4 أو AcrVA5.
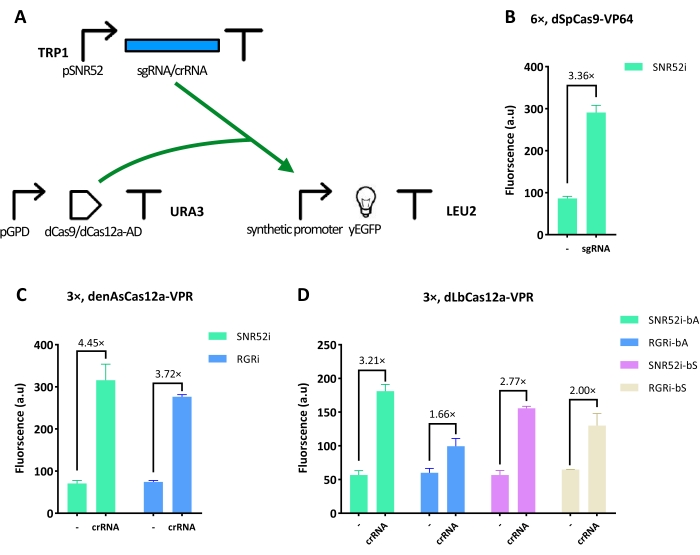
الشكل 1: التنشيط النسخي بوساطة dCas9 / dCas12a-AD. أ: مخطط الدائرة 1. احتوت المروجين التركيبية ل yEGFP على ست (6x) وثلاث (3x) نسخ من المواقع المستهدفة ل dCas9 أو dCas12a ، على التوالي. بعد ذلك ، يتحد dCas9 / dCas12a-AD مع sgRNA / crRNA. يتم استهداف المروج الاصطناعي وتنشيطه بسبب وجود مجالات التنشيط. (ب) تم تحقيق أفضل كفاءة تنشيط ل dSpCas9-VP64 عندما كان هناك ستة مواقع مستهدفة lexOp على المروج الاصطناعي ، وتم نسخ sgRNA بواسطة SNR52i. (ج، د) تم الحصول على أعلى كفاءة تنشيط ل dCas12a-VPR عندما تم إدخال ثلاث نسخ من lexOp في المروج الاصطناعي ، وتم إنشاء crRNA بواسطة SNR52i. SNR52i يعني أن sgRNA / crRNA تم إنتاجه بواسطة pSNR52 ، وتم وضع شريط التعبير داخل متجه مكوك تكاملي. RGRi يعني استخدام بلازميد تكاملي يستضيف شريط RGR للتعبير عن sgRNA / crRNA. يمثل التحكم السلبي ، "-" ، sgRNA / crRNA يحتوي على تسلسل فاصل مخلوط لا يتطابق مع موقع lexOp ولا مع أي تسلسل على طول جينوم الخميرة. يشير "bA" إلى أن sgRNA / crRNA يربط الشريط المضاد للحمض النووي المستهدف ، بينما يشير "bS" إلى ربط الشريط الحسي. يمثل كل مستوى مضان القيمة المتوسطة من ثلاث تجارب مستقلة على الأقل (أي أجريت في أيام مختلفة). أشرطة الخطأ هي الانحراف المعياري للمتوسط. يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
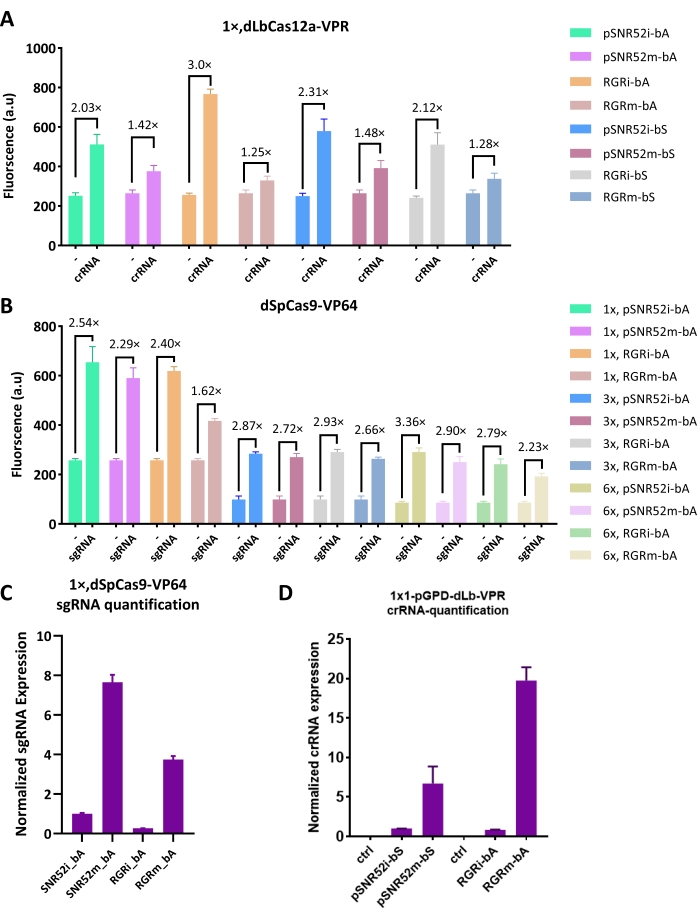
الشكل 2: مقارنة بين sgRNA / crRNA التكاملي والمنتج للبلازميد العرضي. (أ) كفاءة تنشيط dLbCas12a-VPR: crRNA عند استهداف موقع واحد على المروج المنبع ل yEGFP. (B) dSpCas9-VP64: sgRNA تنشيط n× المروج الاصطناعي ("n" تعني عدد المواقع المستهدفة lexOp). (ج، د) مستوى تعبير sgRNA / crRNA الطبيعي15,16. "i" تعني أن شريط تعبير sgRNA / crRNA قد تم وضعه في متجه مكوك تكاملي ، بينما يشير الحرف "m" إلى البلازميد متعدد النسخ (أي العرضي). يشير "bA" / "bS" إلى أن sgRNA / crRNA يربط الشريط المضاد / الحسي للحمض النووي. "ctrl" هو عنصر تحكم سلبي ، حيث تم التعبير عن sgRNA / crRNA المخفوق. يمثل كل مستوى مضان القيمة المتوسطة من ثلاث تجارب مستقلة على الأقل (أي أجريت في أيام مختلفة). أشرطة الخطأ هي الانحراف المعياري للمتوسط. يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
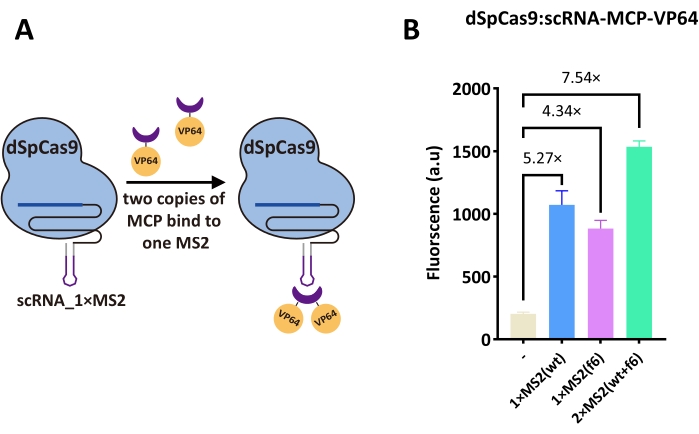
الشكل 3: كفاءة تنشيط dSpCas9 العارية في مجمع مع scRNA. (أ) الرسم التخطيطي للتفاعلات بين scRNA و MCP-VP64 و dSpCas916 العاري. يمثل الهيكل الشبيه بالغطاء باللون الأرجواني MCP (بروتين معطف MS2). يمكن لدبوس شعر MS2 واحد (الهيكل الأرجواني في scRNA) تجنيد وربط نسختين من MCP. وبالتالي ، فإن scRNA لا يتيح فقط ربط الحمض النووي بواسطة dSpCas9 ولكن أيضا تنشيط التعبير الجيني من خلال توظيف MCP-VP64. (ب) كفاءة تنشيط dSpCas9: scRNA-MCP-VP64. تم اختبار ثلاثة أنواع من scRNA: واحد يحمل دبوس الشعر MS2 من النوع البري -1×MS2 (بالوزن) ، وآخر مصمم باستخدام f6 MCP aptamer-1×MS2 (f6) ، والأخير يحتوي على كل من دبابيس الشعر -2×MS2 (wt + f6) ، والتي تبين أنها الأكثر أداء. يمثل كل مستوى مضان القيمة المتوسطة من ثلاث تجارب مستقلة على الأقل (أي أجريت في أيام مختلفة). أشرطة الخطأ هي الانحراف المعياري للمتوسط. يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.

الشكل 4: الدوائر والنتائج المتعلقة ب AcrIIA. (أ) أدخل شريط تعبير AcrIIA في الدائرة 1. تتضمن هذه الوحدة الإضافية pGPD التي تقود التعبير عن AcrIIAs لمواجهة dSpCas9: scRNA_2× MS2 (wt + f6) -MCP-VP64. (ب) كفاءة تثبيط AcrIIAs على أفضل منشط قائم على dSpCas9 في الشكل 3B. يمثل الخط الأسود المتقطع التألق في وجود المنشط المستند إلى dSpCas9. توضح الأشكال الموجودة أعلى كل عمود كفاءة التثبيط المحسوبة على أنها نسبة OFF / ON (أي مستوى التألق في وجود AcrIIA مقسوما على التألق في حالة عدم وجود أي AcrIIA). في الأسطورة ، تزداد قوة المروجين التأسيسيين الأربعة تدريجيا من أعلى إلى أسفل. (ج) رسم تخطيطي لجهاز استشعار β-استراديول (بوابة NOT) يعبر عن AcrIIA4-HBD (hER). د: منحنى معايرة الدائرة في (ج)16. يشير المنحنى الأخضر إلى التغير في التألق في الدائرة الوظيفية. تم اشتقاق المنحنى الأسود من الإجهاد دون التعبير عن AcrIIA4-HBD (hER) - السيطرة السلبية. يشير الخط المتقطع باللون الرمادي إلى هضبة التألق عند التوازن. تم حسابه كمتوسط لقيم التألق بتركيزات β-استراديول لا تقل عن 125 نانومتر. يمثل كل مستوى مضان القيمة المتوسطة من ثلاث تجارب مستقلة على الأقل (أي أجريت في أيام مختلفة). أشرطة الخطأ هي الانحراف المعياري للمتوسط. يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
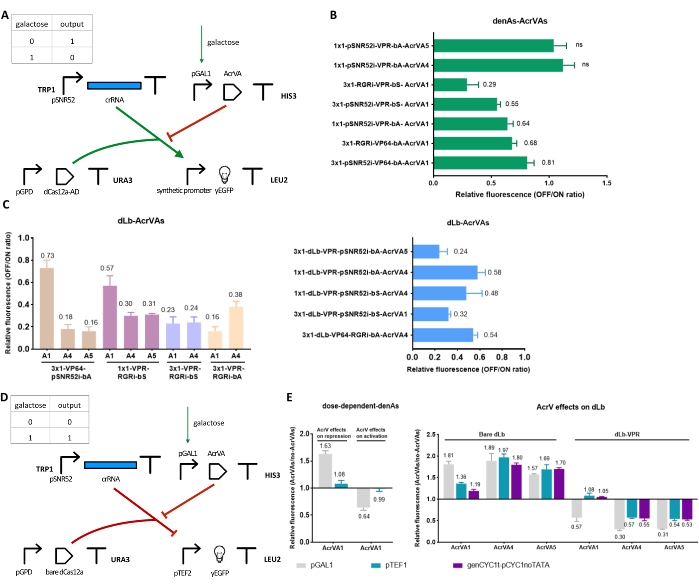
الشكل 5: الدوائر والنتائج المتعلقة ب AcrVA. (أ) أدخل شريط تعبير AcrVA في الدائرة 1. يحتوي TU الجديد على المروج المحرض pGAL1 المنبع لجينات AcrVA التي تحيد عمل dCas12a-AD. الدائرة الجديدة عبارة عن بوابة NOT ينظمها الجالاكتوز. (ب، ج) النتائج من بوابة NOT المستجيبة للجالاكتوز في (A). هنا ، يقود pGAL1 توليف AcrVA ، والتي تتفاعل بعد ذلك مع dCas12a-AD15. يتوافق التألق النسبي مع نسبة OFF / ON. (د) بوابة نعم المستجيبة للغالاكتوز. يستخدم AcrVAs تحت سيطرة pGAL1 و dCas12as العارية التي تقمع تخليق yEGFP. (ه) مقارنة كفاءات تثبيط AcrVA التي عبر عنها المروجون من مختلف القوة15. تشير المجموعات "تأثيرات AcrV على القمع" و "dLb المجردة" إلى الدائرة في (D). المجموعات "تأثيرات AcrV على التنشيط" و "dLb-VPR" هي نتائج بوابة NOT في (A). يمثل كل مستوى مضان القيمة المتوسطة من ثلاث تجارب مستقلة على الأقل (أي أجريت في أيام مختلفة). أشرطة الخطأ هي الانحراف المعياري للمتوسط. يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
الجدول التكميلي 1: قائمة بجميع تسلسلات الحمض النووي المستخدمة في هذه الدراسة. الرجاء الضغط هنا لتنزيل هذا الملف.
الجدول التكميلي 2: قائمة الاشعال المستخدمة في هذه الدراسة. الرجاء الضغط هنا لتنزيل هذا الملف.
ملفات الترميز التكميلية: البرنامج النصي R studio لتحليل ملفات FCS. الرجاء الضغط هنا لتنزيل هذا الملف.
Discussion
أظهر البروتوكول سير عمل كامل محتمل للدوائر الرقمية للجينات الاصطناعية ، بعد دورة الهندسة البيولوجية "Design-Build-Test-Learn" (DBTL) وفيما يتعلق بكل من تجارب المختبر الجاف والمختبر الرطب. هنا ، ركزنا على نظام CRISPR-Cas ، بشكل أساسي dSpCas9 و denAsCas12a و dLbCas12a والبروتينات المضادة ل CRISPR المقابلة ، من خلال تصميم وبناء شبكات النسخ الصغيرة S. cerevisiae. بعضها يحاكي البوابات المنطقية ، وهي المكونات الأساسية للدوائر الرقمية. سمحت لنا جميع الدوائر الموصوفة هنا بتصوير خصائص وميزات البروتينات المرتبطة بكريسبر والمضادة لكريسبر في S. cerevisiae. هذه النتائج ضرورية لتضمين هذه البروتينات في مخطط الدوائر الرقمية الجينية.
يوفر مفهوم DBTL إطارا في البيولوجيا التركيبية ، بينما يجب إجراء العديد من التحسينات والتحسينات بعد اختبار قطعة أثرية جديدة. على سبيل المثال ، في الدائرة 1 ، كان هناك في البداية موقع هدف واحد فقط (نسخة واحدة من lexOp) ل dCas9 / dCas12a-AD على المروج الاصطناعي في المنبع من yEGFP. بعد اختبار تكوين الدائرة هذا ، وجدنا أنه لا يمكن أن يحقق أكثر من تنشيط مزدوج15,16. بعد ذلك ، افترضنا أنه من خلال زيادة عدد نسخ lexOp ، كما في7 ، يمكننا الوصول إلى تنشيط نسخ أعلى. في الواقع ، تم الحصول على مستوى مضان أعلى باستخدام ثلاثة إلى ستة مواقع lexOp (الشكل 1). علاوة على ذلك ، قمنا بتحسين أداء الدوائر التي تستضيف dSpCas9 من خلال هندسة scRNA ، وهو أسهل من دمج واحد أو أكثر من ADs مع بروتين كبير مثل dSpCas9 (الشكل 3). بالإضافة إلى ذلك ، باستخدام مروج لنقاط قوة مختلفة لإنتاج AcrIIAs الثلاثة التي اخترناها ، استنتجنا أن AcrIIA4 كان أقوى مثبط بينها. وبالتالي ، قمنا ببناء بوابة NOT جديدة تستجيب ل β-estradiol عن طريق دمج HBD (ER) إلى AcrIIA4 واستغلال القمع القوي ل AcrIIA4 على أفضل منشط قائم على dSpCas9 (الشكل 4).
وبالمثل ، قمنا بتمييز كل من عمل denAsCas12a و dLbCas12a بعمق في الخميرة وتفاعلاتها مع ثلاثة AcrVA (الشكل 5). لكل زوج من dCas12a-AcrVA ، قمنا ببناء NOT (تم دمج dCas12a في AD) وبوابة YES (bare dCas12a) تستجيب للجالاكتوز. بشكل عام ، نتج عن dLbCas12a ، جنبا إلى جنب مع AcrVA5 ، أفضل نظام لحساب وظائف المنطق البسيطة.
قدمت الطريقة الموضحة هنا بعض الخطوات الحاسمة. كانت جميع تسلسلات الحمض النووي للبروتين محسنة لكودون الخميرة لضمان تعبير أعلى في S. cerevisiae. لتجنب الأهداف غير المحددة ل dSpCas9 / denAsCas12a / dLbCas12a في جينوم S. cerevisiae ، اخترنا عاملا بكتيريا مثل lexOp. علاوة على ذلك ، أظهرت السلالات التي تحتوي على محفز GAL1 تأخيرا كبيرا في النمو يمكن أن يحد من قابلية تطبيق pGAL1 على دوائر الجينات الاصطناعية15.
يمكن أيضا إدخال بعض التعديلات على بعض الخطوات في الطريقة الشاملة. من أجل تحسين كفاءة إجراء ربط الهضم ، من الأفضل هضم 10 ميكروغرام (بين عشية وضحاها) من كل من البلازميد المحتوي على الإدراج وناقلات المستقبل ، بدلا من 5 ميكروغرام فقط في 1 ساعة. بهذه الطريقة ، يتم الوصول إلى تركيز أعلى للحمض النووي بعد خطوة الشطف. يجب تمديد وقت ربط T4 من 1 ساعة (بروتوكول الشركة المصنعة) إلى 8 ساعات. وأخيرا، ينبغي تخفيف السلالات التي تحتوي على بروتين الاندماج dCas12a-VPR بعد استزراع لمدة 24 ساعة وزراعتها لمدة 12 ساعة أخرى قبل إجراء تجربة نظام مراقبة الأصول الميدانية. في ظل هذه الحالة ، لم يعد التباين بين مستويات التألق من الخلايا المختلفة مرتفعا جدا ، ويرافق الانحراف المعياري المقبول القيمة المتوسطة لشدة التألق على مجموعة الخلايا.
باختصار ، شرح هذا البروتوكول كيفية تبسيط تصميم الدوائر الرقمية الجينية من خلال الاستفادة من بروتينات dCas ، وربما البروتينات المضادة لكريسبر. والأهم من ذلك ، أظهرنا بالتفصيل كيف تعمل عائلات البروتينات هذه في S. cerevisiae وأي منها واعد للاستخدام المستقبلي داخل الشبكات الرقمية. المشكلة التي لم يتم حلها هي اقتران أنظمة CRISPR-dCas / المضادة ل CRISPR والمواد الكيميائية ، والتي تمثل مدخلات الدائرة ولا يمكنها ربط بروتينات dCas أو مضادات CRISPR مباشرة. هنا ، تجاوزنا المشكلة باستخدام إما مروج GAL1 المستحث أو HBD (ER) المرفق ب AcrIIA4. ومع ذلك ، فإن طريقة لتعميم بنية طبقة إدخال الدائرة ضرورية لتصميم دوائر رقمية للجينات الاصطناعية لمجالات الهندسة الحيوية المختلفة مثل الهندسة الأيضية ، والتخليق الحيوي ، والاستشعار الحيوي ، والتشخيص الحيوي ، والمعالجة الحيوية.
Disclosures
يعلن أصحاب البلاغ عدم وجود مصلحة مالية متنافسة.
Acknowledgements
نود أن نشكر جميع طلاب مختبر البيولوجيا الاصطناعية - SPST ، TJU - على مساعدتهم العامة ، جنبا إلى جنب مع Zhi Li و Xiangyang Zhang لمساعدتهم في تجارب FACS.
Materials
| Name | Company | Catalog Number | Comments |
| 0.1 mL PCR 8-strip tubes | NEST | 403112 | |
| 0.2 mL PCR tubes | Axygen | PCR-02-C | |
| 1.5 mL Microtubes | Axygen | MCT-150-C | |
| 15 mL Centrifuge tubes | BIOFIL | CFT011150 | |
| 2 mL Microtubes | Axygen | MCT-200-C | |
| 50 mL Centrifuge tubes | BIOFIL | CFT011500 | |
| Agarose-molecular biology grade | Invitrogen | 75510-019 | |
| Ampicillin sodium salt | Solarbio | 69-52-3 | |
| Applied biosystems veriti 96-well thermal cycler | Thermo Fisher Scientific | 4375786 | |
| AxyPrep DNA gel extraction kit | Axygen | AP-GX-250 | |
| BD FACSuite CS&T research beads | BD | 650621 | Fluorescent beads |
| BD FACSVerse flow cytometer | BD | - | |
| Centrifuge | Eppendorf | 5424 | |
| Centrifuge Sorvall ST 16R | Thermo Fisher Scientific | 75004380 | |
| E. coli competent cells (Strain DH5α) | Life Technologies | 18263-012 | |
| ECL select Western Blotting detection reagent | GE Healthcare | RPN2235 | |
| Electrophoresis apparatus | Beijing JUNYI Electrophoresis Co., Ltd | JY300C | |
| Flat 8-strip caps | NEST | 406012 | |
| Gene synthesis company | Azenta Life Sciences | https://web.azenta.com/zh-cn/azenta-life-sciences | |
| Goat anti-Mouse IgG (H+L) cross-adsorbed secondary antibody Alexa Fluor 568 | Invitrogen | A-11004 | |
| HiFiScript cDNA synthesis kit | CWBIO | CW2569M | Kit used in step 6.2.2.1 |
| Lysate solution (Zymolyase) | zymoresearch | E1004-A | |
| Nikon Eclipse 80i fluorescence microscope | Nikon | - | Fluorescence microscope |
| Pipet tips—10 μL | Axygen | T-300-R-S | |
| Pipet tips—1000 μL | Axygen | T-1000-B-R-S | |
| Pipet tips—200 μL | Axygen | T-200-Y-R-S | |
| pRSII403 | Addgene | 35436 | |
| pRSII404 | Addgene | 35438 | |
| pRSII405 | Addgene | 35440 | |
| pRSII406 | Addgene | 35442 | |
| pRSII424 | Addgene | 35466 | |
| pTPGI_dSpCas9_VP64 | Addgene | 49013 | |
| Q5 High-fidelity DNApolymerase | New England Biolabs | M0491 | |
| Restriction enzyme-Acc65I | New England Biolabs | R0599 | |
| Restriction enzyme-BamHI | New England Biolabs | R0136 | |
| Restriction enzyme-SacI-HF | New England Biolabs | R3156 | |
| Restriction enzyme-XhoI | New England Biolabs | R0146 | |
| Roche LightCycler 96 | Roche | - | Real-Time PCR Instrument |
| S. cerevisiae CEN.PK2-1C | - | - | The parent strain. The genotype is: MATa; his3D1; leu2-3_112; ura3-52; trp1-289; MAL2-8c; SUC2 |
| Stem-Loop Kit | SparkJade | AG0502 | Kit used in step 6.2.1.3 |
| T100 Thermal Cycler | BIO-RAD | 186-1096 | |
| T4 DNA ligase | New England Biolabs | M0202 | |
| T5 Exonuclease | New England Biolabs | M0363 | |
| Taq DNA ligase | New England Biolabs | M0208 | |
| Taq DNA polymerase | New England Biolabs | M0495 | |
| TB Green Premix Ex Taq II (Tli RNaseH Plus)(2x) (SYBR Green I dye) | Takara | RR820Q | |
| YeaStar RNA kit | Zymo Research | R1002 | |
| β-estradiol | Sigma-Aldrich | E8875 |
References
- Marchisio, M. A., Stelling, J. Automatic design of digital synthetic gene circuits. PLOS Computational Biology. 7 (2), 1001083 (2011).
- Karnaugh, M. The map method for synthesis of combinational logic circuits. Transactions of the American Institute of Electrical Engineers. 72 (9), 593-599 (1953).
- Mandell, J. G., Barbas, C. F. Zinc finger tools: custom DNA-binding domains for transcription factors and nucleases. Nucleic Acids Research. 34, 516-523 (2006).
- Bogdanove, A. J., Voytas, D. F. TAL effectors: customizable proteins for DNA targeting. Science. 333 (6051), 1843-1846 (2011).
- Drinnenberg, I. A., et al. RNAi in budding yeast. Science. 326 (5952), 544-550 (2009).
- Gander, M. W., Vrana, J. D., Voje, W. E., Carothers, J. M., Klavins, E. Digital logic circuits in yeast with CRISPR-dCas9 NOR gates. Nature Communications. 8, 15459 (2017).
- Farzadfard, F., Perli, S. D., Lu, T. K. Tunable and multifunctional eukaryotic transcription factors based on CRISPR/Cas. ACS Synthetic Biology. 2 (10), 604-613 (2013).
- Nakamura, M., et al. Anti-CRISPR-mediated control of gene editing and synthetic circuits in eukaryotic cells. Nature Communications. 10 (1), 194 (2019).
- Louvion, J. F., Havaux-Copf, B., Picard, D. Fusion of GAL4-VP16 to a steroid-binding domain provides a tool for gratuitous induction of galactose-responsive genes in yeast. Gene. 131 (1), 129-134 (1993).
- DiCarlo, J. E., et al. Genome engineering in Saccharomyces cerevisiae using CRISPR-Cas systems. Nucleic Acids Research. 41 (7), 4336-4343 (2013).
- Gao, Y., Zhao, Y. Self-processing of ribozyme-flanked RNAs into guide RNAs in vitro and in vivo for CRISPR-mediated genome editing. Journal of Integrative Plant Biology. 56 (4), 343-349 (2014).
- Jinek, M., et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337 (6096), 816-821 (2012).
- Zetsche, B., et al. Cpf1 is a single RNA-guided endonuclease of a class 2 CRISPR-Cas system. Cell. 163 (3), 759-771 (2015).
- Fonfara, I., Richter, H., Bratovic, M., Le Rhun, A., Charpentier, E. The CRISPR-associated DNA-cleaving enzyme Cpf1 also processes precursor CRISPR RNA. Nature. 532 (7600), 517-521 (2016).
- Yu, L., Marchisio, M. A. Saccharomyces cerevisiae synthetic transcriptional networks harnessing dCas12a and Type V-A anti-CRISPR proteins. ACS Synthetic Biology. 10 (4), 870-883 (2021).
- Zhang, Y., Marchisio, M. A. Interaction of bare dSpCas9, scaffold gRNA, and type II anti-CRISPR proteins highly favors the control of gene expression in the yeast S. cerevisiae. ACS Synthetic Biology. 11 (1), 176-190 (2022).
- Sheff, M. A., Thorn, K. S. Optimized cassettes for fluorescent protein tagging in Saccharomyces cerevisiae. Yeast. 21 (8), 661-670 (2004).
- Naito, Y., Hino, K., Bono, H., Ui-Tei, K. CRISPRdirect: software for designing CRISPR/Cas guide RNA with reduced off-target sites. Bioinformatics. 31 (7), 1120-1123 (2015).
- Chee, M. K., Haase, S. B. New and redesigned pRS plasmid shuttle vectors for genetic manipulation of Saccharomyces cerevisiae. G3: Genes|Genomes|Genetics. 2 (5), 515-526 (2012).
- Gibson, D. G. Synthesis of DNA fragments in yeast by one-step assembly of overlapping oligonucleotides. Nucleic Acids Research. 37 (20), 6984-6990 (2009).
- Froger, A., Hall, J. E. Transformation of plasmid DNA into E. coli using the heat shock method. Journal of Visualized Experiments. (6), e253 (2007).
- Green, M. R., Sambrook, J. . Molecular Cloning. Fourth edition. , (2012).
- Sanger, F. Determination of nucleotide sequences in DNA. Science. 214 (4526), 1205-1210 (1981).
- Gietz, R. D., Woods, R. A. Transformation of yeast by lithium acetate/single-stranded carrier DNA/polyethylene glycol method. Methods in Enzymology. 350, 87-96 (2002).
- Zalatan, J. G., et al. Engineering complex synthetic transcriptional programs with CRISPR RNA scaffolds. Cell. 160 (1-2), 339-350 (2015).
- Song, W., Li, J., Liang, Q., Marchisio, M. A. Can terminators be used as insulators into yeast synthetic gene circuits. Journal of Biological Engineering. 10, 19 (2016).
- Rauch, B. J., et al. Inhibition of CRISPR-Cas9 with bacteriophage proteins. Cell. 168 (1-2), 150-158 (2017).
- Hynes, A. P., et al. An anti-CRISPR from a virulent streptococcal phage inhibits Streptococcus pyogenes Cas9. Nature Microbiology. 2 (10), 1374-1380 (2017).
- Watters, K. E., Fellmann, C., Bai, H. B., Ren, S. M., Doudna, J. A. Systematic discovery of natural CRISPR-Cas12a inhibitors. Science. 362 (6411), 236-239 (2018).
- Chen, C., et al. Real-time quantification of microRNAs by stem-loop RT-PCR. Nucleic Acids Research. 33 (20), 179 (2005).
- Pfaffl, M. W. A new mathematical model for relative quantification in real-time RT-PCR. Nucleic Acids Research. 29 (9), 45 (2001).
- Hahne, F., et al. flowCore: a Bioconductor package for high throughput flow cytometry. BMC Bioinformatics. 10 (1), 106 (2009).
- Li, J., Xu, Z., Chupalov, A., Marchisio, M. A. Anti-CRISPR-based biosensors in the yeast S. cerevisiae. Journal of Biological Engineering. 12, 11 (2018).
- Dong, L., et al. An anti-CRISPR protein disables type V Cas12a by acetylation. Nature Structural & Molecular Biology. 26 (4), 308-314 (2019).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved